嘉峪检测网 2024-10-10 18:08
导读:我们讨论了合成生物学使能技术主流中各种原理和技术的进步,包括基因组的合成与组装、DNA存储、基因编辑、分子进化和功能蛋白的从头设计等。
摘要:合成生物学为生命科学研究提供了一种新的范式("构建以学习"),并开启了生物技术的未来之旅("构建以使用")。在这里,我们讨论了合成生物学使能技术主流中各种原理和技术的进步,包括基因组的合成与组装、DNA存储、基因编辑、分子进化和功能蛋白的从头设计、细胞与基因回路工程、无细胞合成生物学、人工智能(AI)辅助合成生物学,以及生物铸造厂。我们还介绍了定量合成生物学的概念,它正在引导合成生物学朝着更高的准确性和可预测性或真正的合理设计方向发展。我们得出结论,合成生物学将通过使能技术的迭代发展和核心理论的成熟,建立其学科体系。
1.引言
合成生物学,亦称为工程生物学,是一门新兴的跨学科学科。它整合了生物科学、化学、物理、材料科学、计算机和信息科学以及工程概念,重新设计或从头设计和构建生物系统,创造了一种被称为“构建以学习”的生物研究新范式,并赋予当前生物技术一种新的动力,称为“构建以使用”。合成生物学由三个相互关联的方面组成:理论、使能技术以及应用研究。
半个多世纪前,在理解了核酸和蛋白质的基本结构后,科学家们实现了从化学合成小分子到化学合成生物大分子的历史性飞跃。开创性的研究包括结晶牛胰岛素的全合成、遗传密码的多核苷酸合成以及氨基酸转移酶 RNA。世纪之交,随着人类基因组计划的完成以及 DNA 测序和合成技术的进步,合成生物学实现了从核酸合成到基因组合成的新飞跃。从简单到复杂,科学家们已经合成了病毒基因组、细菌基因组以及酵母染色体。这些里程碑式的方法为生物系统的合成提供了基础,证明了合成基因组可以完全执行自然生物功能。
在同一时期,工程概念被引入到生物系统的设计和创造中,例如基因电路、生物装置和模块、最小基因组和底盘细胞。这些概念通过许多人工逻辑生物装置和基因组得到了很好的解释,例如基因开关、合成转录调节因子的振荡网络、基于群体感应的细胞间通信电路、用于程序化模式形成的合成多细胞系统、执行逻辑运算的 RNA 装置以及可编程的微生物杀伤开关。这些开创性的探索旨在实现生物组件的标准化、底盘细胞的通用化,以及基于工程原理设计生物系统的可预测性。
然而,由于生物系统的遗传变异、代谢多样性和动态性以及生物量的灵活性,它们是极其复杂的系统。我们为生物设计所获得的知识远远不足以满足工程标准。高通量、多周期和自动化的“试错”生物铸造厂应运而生,其效率正通过嵌入人工智能(AI)而得到极大提升。特别是,DeepMind 的 AlphaFold和 Baker 实验室的 RoseTTAFold等高级算法已经彻底改变了蛋白质 3D 结构的预测。这意味着 AI 辅助的蛋白质从头设计将会蓬勃发展。
结果,我们可以区分两种运作方式:数据驱动的“黑箱”和知识驱动的“白箱”。大量的知识输入和数据输出使我们能够进行计算建模,实现生物设计的准确性和可预测性。这种建模被称为定量合成生物学,它正成为合成生物学理论的核心部分。其有效性和可靠性已在研究细菌在新可用栖息地的定殖能力和阐述大肠杆菌生长与细胞周期之间的定量关系中得到证明。
2.定量合成生物学
2.1.合成生物学研究的层次结构
生命系统的复杂性相当大,这是由于它们的层次组织和相互作用层级(图1A)。复杂系统所表现出的特性通常被称为“涌现特性”,这是由于组成部分之间的相互作用而产生的,即使完全了解所有部分,也无法预测。例如,识别一个生物体中的基因和蛋白质就像列出飞机的所有部件一样,这并不足以理解飞机是如何飞行的。因此,理解生命属性如何在层次组织中涌现仍然是生命科学中最基本问题之一。
利用生物学和工程原理,合成生物学旨在设计生物模块/反应/系统以实现所需的功能/产品。从生物分子工程、基因组工程到人造细胞设计,合成生物学家可以在任何层次上访问层次生物系统。在每个层次上,合成生物学家的目标是创造一个更合成和复杂的入口,因此合成生物学的进步通常导致最复杂和最不自然的系统。总的来说,合成生物学研究生命属性如何在层次化的生命系统中涌现,采用自下而上的策略,即“通过构建来理解”。
正如理查德·P·费曼(Richard P. Feynman)著名的论断:“我们不能创造的,我们就不能理解。”
合成生物学已被证明是探索生命属性所有三个维度的有价值工具,即以前存在过的功能、现在存在和尚未存在过的功能。它允许创造人工系统,如基于硅的生命和单染色体酵母,以探索生命的边界并揭示“生命是什么”的基本问题。相比之下,这些人工系统可以超越自然细胞的能力,这将极大地促进生物技术的发展,以改变我们的日常生活,即探索不存在的生命功能。通过工程/重建生物系统,合成生物学使我们能够从传统生物学中提取不可接近的信息,并帮助我们理解生命原理(现有功能)。最后,合成生物学家通过简化和建模研究复杂生物系统。这些努力推进了我们对其底层物理的理解,这将有助于阐明早期地球环境中可能的进化路径(现有功能)。
尽管在理解生命系统方面取得了相当大的进展,但仍然缺少一种成熟的合成生物学方法。为了解决这个问题,我们首先总结了以前系统的一般范式。一个综合生物学研究实践通常包括三个层次,称为部分、拓扑和功能。功能来自部分,但在复杂系统中,很难获得它们之间的端到端关系。因此,增加了一个拓扑中间层。拓扑如何从部分中涌现被称为机制,功能如何从拓扑中涌现被称为原理(图1B)。原理更通用,而机制特定于所使用的部分。例如,艾伦·图灵(Alan Turing)提出了反应-扩散原理,这在自然界广泛调节模式形成。基于这一通用原理,研究人员构建了化学Belousov-Zhabotinsky反应、生物昼夜节律振荡和工程DNA/酶反应网络的人工反应-扩散系统,以遵循它们各自的机制。因此,理解这些原理为合理设计奠定了基础,这反过来加速了合成生物学向增加复杂性和效率的方向发展,推进了我们对生命系统基本原理的理解(图1C)。我们称之为合理设计的理想合成生物学实践,是基于兴趣原理设计基本组件以实现预期功能。
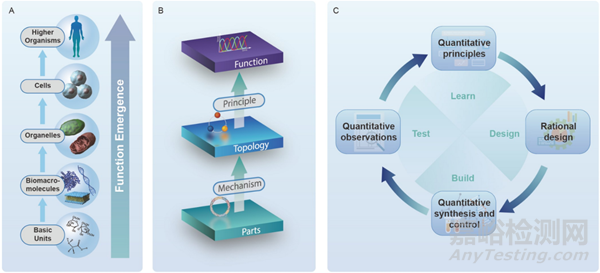
图1 通过定量合成生物学理解功能出现。A,在生命系统的层次组织中出现的功能,仅通过了解单个部分是无法理解的。B,方法论的层次结构。C,定量合成生物学研究中的原则和合理设计相互促进。所提出的定量合成生物学研究范式整合了合理设计、构建和测试。
2.2.定量合成生物学:研究功能涌现的研究范式
成功的合理设计仅限于少数特征明确的系统,例如早期研究中的切换开关和振荡器。近期的例子包括通过形态素介导的人工细胞分化以及能够实现光动力二氧化碳固定的人工光合作用系统。然而,感兴趣的生物学功能的原则常常难以捉摸。在这种情况下,研究人员可以根据对感兴趣功能的定量分析设计新的原则,然后进行测试;或者他们可以依赖于定义明确的组件并进行微调以探索其潜在功能。因此,当一个功能的合理性未知时,合成生物学研究中的常规工作涉及繁琐的试错和在微调中的运气。尽管这种试错研究范式在产生关键信息和扩展我们对复杂生物系统的理解方面取得了成功,但在未来几十年中,合成生物学的合理设计将有很高的需求,以有效探索生命系统的基本法则。
我们如何在没有感兴趣原则的情况下实现合理设计?合理设计一个系统是为了可预测性而设计,即基于输入组件和参数预测结果的能力。这要求我们量化自然现象,使我们摆脱定性描述的模糊性和主观性,允许我们发展理论和进行预测。有两种模型指导我们量化自然系统:知识驱动的“白盒”模型和数据驱动的“黑盒”模型。白盒模型是基于宏观实验观察建立的。通过综合分析,我们可以将这些复杂的观察以数学公式描述,从而提取一般的理论框架和基本原理。例如,刘等人开发了一个由多个偏微分方程组成的反应-扩散模型,描述了细菌菌落范围扩展系统中的细胞生长、细胞移动等,再现了该系统中自发出现的时空模式;郑等人发现细胞质量和染色体复制-分离速率之间存在线性关系,为理解细胞周期调控和编程细胞大小提供了定量基础。特伦斯·华的团队制定了一些细菌生长定律,并建立了蛋白质组资源分配的原则,为理解细菌对生理扰动的响应和设计合成代谢途径提供了预测模型。相比之下,黑盒建模侧重于输入和输出之间的直接相关性。大量已知的输入和输出数据将被用来训练和改进算法,然后可以用来预测相关系统的结果。例如,DeepMind开发的AlphaFold成功预测了98.5%的人类蛋白质结构,这些是基于已知的氨基酸序列-蛋白质结构关系训练的。
白盒和黑盒建模都被证明是生物设计中的有价值工具:通过开发机制模型和系统分析模型,周等人识别了能够实现自发细胞极化的网络拓扑结构。根据模型的预测,他们成功构建了在酵母中产生极化蛋白质分布的合成基因回路。陆等人开发了一种机器学习算法,预测PET(一种主要类型的塑料)水解酶如何突变以提高它们的效率和鲁棒性。在该算法的指导下,团队对野生型酶进行了工程改造,并获得了一种具有更优越PET降解活性的突变体。尽管采取了不同的途径,但两种方法都旨在通过定量关系构建自然现象,实现预测性设计。因此,我们提出了定量合成生物学的概念,作为当前合理设计瓶颈的紧迫研究范式。
定量合成生物学是定量生物学和合成生物学的交叉点。它从底层研究合成生物学系统,并使用简化的定量关系描述复杂的生物现象。在白盒和黑盒建模的指导下,可以获得对生命系统的定量理解,从而我们可以发展复杂生物系统的理论并探索它们的基本法则。对基本原理的理解将促进合理设计,从而加速实现真正的合成生物学工程。白盒和黑盒建模都涉及大量数据,这可以通过建立自动化和高通量的实验设施以及标准化的协议、算法和工作流程(测试)来实现。最后,应进一步发展使能技术以精确控制/重建生物系统,如高效准确的DNA合成技术、基因组编辑、基因回路设计和蛋白质定向进化(构建)。因此,我们提出了合成生物学的未来研究范式,包括设计、构建和测试(图1C)。我们设想这个研究周期将彻底改变当前定性、描述性和有限的合成生物学研究,进入一个具有定量、理论和系统特征的新阶段。这场革命将通过回答关于生命功能如何运作的基本问题,推动生物学的前沿,这反过来将帮助我们设计具有更好预测能力的合成系统。
3.合成和组装基因组
3.1.基因组合成的简史
合成基因组学的历史可以追溯到1970年,当时经过5年的努力合成了77个碱基对的酵母tRNA基因。2002年,化学构建了一个7.5 kb的脊髓灰质炎病毒互补DNA。一年后,仅用两周时间就创造了5.5 kb的X174噬菌体基因组。受到这一成功的鼓舞,一些研究小组开始构建583 kb的Mycoplasma genitalium JCVI-1.0基因组,并于2008年完成。随后,合成了1.1 Mbp的Mycoplasma mycoides JCVI-syn1.0基因组并证明其功能。到目前为止,合成基因组大多模仿了自然模板DNA。2016年,科学家们使用三个设计-构建-测试周期将1.1 Mbp的JCVI-syn1.0基因组最小化为功能性的531 kbp JCVI-syn3.0。除评估基因内容的可塑性外,基因组合成还允许重新编程遗传密码。2016年,设计并实验验证了一个3.97兆碱基、57个密码子的大肠杆菌基因组,占合成基因组的63%。三年后,创建了一个具有61个密码子的合成基因组的大肠杆菌变体,实现了整个基因组意义密码子的首次压缩(图2)。在合成病毒和细菌基因组的同时,启动了一个名为合成酵母基因组项目(Sc2.0)的尝试,该项目现在接近完成,努力合成整个酿酒酵母基因组(约12 Mb,分为16条染色体),并进行了许多改变以探索有关基因组功能的基本生物学问题。2016年,提出了一个更雄心勃勃的项目,基因组编写项目(GPW),以重写千兆碱基大小的复杂基因组。然而,当前的DNA合成能力和成本是构建如此大基因组的主要限制因素,因此迫切需要在DNA合成和基因组组装方面取得突破。
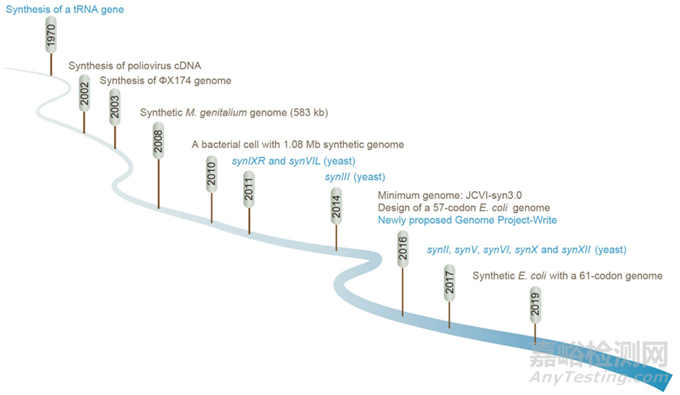
图2 合成基因组学里程碑。棕色代表在病毒和细菌基因组合成方面的进展。蓝色表示在真核生物基因组合成方面的里程碑。
3.2.基因合成和基因组组装的技术发展
3.2.1.寡核苷酸合成
目前,最常用的寡核苷酸合成技术是20世纪80年代开发的固相磷酸酰胺化学合成方法。在这种方法中,每个核苷酸单体的添加通过去保护、偶联、加帽和氧化的四个步骤循环进行。然后通过去除保护基团,为下一个碱基重复该循环。这种方法的稳健性和保真性使其能够自动化和工业化。自20世纪90年代以来,基于这种方法的DNA合成器已经被开发出来,同时合成96-1536个不同的寡核苷酸。相比之下,阵列基础的寡核苷酸合成技术理论上可以显著降低成本并提高产量。然而,作者指出,由于逐步多重反应系统中不可避免的合成相关错误,合成质量通常随着寡核苷酸长度的增加而降低。尽管不断努力优化合成过程,合成的寡核苷酸通常不超过200 nt的长度。
酶促从头合成寡核苷酸,早在20世纪60年代就被提出,由于化学合成的长度限制和危险废物问题,已成为一个有希望的替代方案。目前,末端脱氧核糖核苷酸转移酶(TdT)是最佳选择。经过多年的努力,据报道酶促合成可以产生约300个单体,超越了化学合成。迄今为止,已有几家公司成立,以推进酶促DNA合成的商业化。
实现未来快速、高通量按需合成长的DNA分子将大大加速系统生物学中的设计-构建-测试周期。
3.2.2.基因合成
基因合成中的“基因”一词指的是一个长的双链DNA序列,而不是经典定义的基因。商业合成的基因通常在200到3000个碱基对之间。互补的重叠单链寡核苷酸是组装这种双链合成DNA的原料。早期的方法是基于连接的,其中相邻的寡核苷酸通过DNA连接酶进行酶促连接。自从20世纪80年代聚合酶链反应(PCR)发明以来,PCR介导的方法已被广泛用于从寡核苷酸组装所需的DNA序列。此外,Gibson及其同事开发了体外和体内一步法,用于将寡核苷酸直接组装和克隆到质粒中。目前,上述方法已经迭代改进,并在大多数商业和学术应用中使用。此外,由于需要便宜的合成DNA,还开发了使用基于微阵列的寡核苷酸池进行基因合成的方法。
除了基因,各种应用需要超过10 kb甚至100 kb的更长DNA分子,这导致了开发一系列组装短DNA的方法,如BioBrick、BglBrick、iBrick和HVAS。然而,这些基于内切酶的技术产生的“疤痕”序列可能会影响最终构建的功能或引入不需要的变异。II型限制性内切酶的特点是切割位点仅在识别位点的几个碱基之外,使它们成为“无疤痕”组装的理想解决方案。基于这一原理,开发了Golden Gate方法和工具包,并获得了显著的流行。此外,为了消除对限制性内切酶的需求,已经建立了几种无缝组装方法,如Gibson组装、连接酶循环反应、序列和连接独立克隆、循环聚合酶扩展克隆和酵母组装。目前,使用哪种组装技术是一个偏好问题。重要的是,上述大多数方法都可以自动化,以增加构建长合成DNA的通量。
3.2.3.基因组组装
要合成小基因组,限制性克隆或聚合酶循环组装(PCA)方法通常足够,而不同工具的组合被用于构建更大规模(超过100千碱基)的合成染色体或基因组。
尽管Gibson组装已被报道可以组装高达几百千碱基的DNA分子,体外过程的效率随着DNA长度的增加而下降,使其主要成为构建几十千碱基合成DNA的商业化工具。相比之下,酵母组装的上限似乎要高得多。除了在100 kb以内或左右的DNA组装高效率之外,一锅酵母转化产生了几百千碱基甚至超过1 Mb的几个合成基因组,如来自5个片段的两个Phaeodactylum tricornutum染色体(497 kb和441 kb),使用25-25 kb片段生成的583 kb细菌基因组,使用16个兆段(38-65 kb大小)的786 kb Caulobacter ethensis-2.0和通过11个100 kb重叠中间体产生的1.08 Mb JCVI-syn1.0基因组。酵母同源重组对于组装Sc2.0中的合成染色体也至关重要,这些染色体与其自身基因组高度相似。常规克隆、Golden Gate、Gibson组装或酵母组装的组合被用来生成“大块”(30-60 kb),这些通过称为逐步切换营养缺陷以促进整合(SwAP-In)的策略顺序引入酵母中,以替换它们的天然对应物。总的来说,这些结果突出了酵母宿主在DNA吸收和组装方面的巨大能力。所有16个酵母染色体都可以重新组织成单一的线性或圆形染色体,这表明酿酒酵母可能能够构建超过10 Mb的DNA分子。
除了酿酒酵母,枯草杆菌、鼠伤寒沙门氏菌和大肠杆菌是另外三个体内基因组组装的宿主。通过“尺蠖”方法,一个3.5 Mb的基因组已被组装进枯草杆菌基因组中。使用枯草杆菌基因组(BGM)载体,一个16.3-kb的小鼠线粒体基因组和一个134.5-kb的水稻叶绿体基因组已成功通过同源重组整合到枯草杆菌基因组中。通过称为滚动圆周扩增片段逐步整合(SIRCAS)的过程,一个200 kb的鼠伤寒沙门氏菌片段被合成DNA替换。在大肠杆菌中,一种基于接合的策略,结合重复复制子执行以通过程序化重组增强基因组工程(REXER),使~4 Mb的重编码基因组得以合成。
3.3.合成基因组学的展望
自下而上的基因组合成能够同时整合密集和复杂的全基因组变化。合成基因组学不仅在生命科学中提供了宝贵的发现。而且还可能引发食品、医疗和化学生产领域的新工业革命。例如,合成病毒已经改变了疫苗的设计和生产,合成基因组正在通过用于移植的人猪器官来拯救生命。然而,目前基因合成的成本对于数百万个碱基对或更长的基因组来说仍然是高不可攀的。此外,还有一些技术障碍需要解决。首先,目前设计的基因组通常在大肠杆菌或酿酒酵母等微生物宿主中组装;然而,某些DNA序列对宿主的毒性常常导致组装失败。其次,长DNA片段的逐步组装受到序列重复性的限制,例如高等真核生物中的中心粒和端粒。第三,将组装好的DNA片段从宿主转移到目标生物体仍然具有挑战性。目前,只有Mycoplasma的圆形基因组已成功移植。最近在高通量DNA合成和组装技术上的发展应该会大大加快合成基因组的构建。使用高密度微芯片、酶促DNA合成和利用微流控技术自动化基因组装的新DNA合成技术的出现将继续降低基因合成的成本。除了在体内组装大的DNA片段,还将出现新的体外合成和放大大DNA片段的技术。在未来五年内,预计基因合成的成本将达到1 Mb以上DNA长度的0.001美元/碱基对。与此同时,大约1 Mb大小的染色体可以在体外完全合成,转移到目标生物体并重新启动宿主,开启合成基因组学的新纪元。与基因组组装相关的一个研究方向是基因组简化,其目标是确定一个活生物的最小基因组。例如,克雷格·文特的团队移除了将近一半的结核菌基因组,以研究细胞存活所需的基因组组成。基于合成染色体重排和LoxPsym介导的进化(SCRaMbLE)的基因组压缩策略揭示了至少60%的合成染色体臂(synXIIL)上的基因对酿酒酵母的细胞活性是可有可无的(Luo等人,2021)。这些研究大大改善了构建具有最小基因组和理想属性的微生物底盘的可行性。未来对多个合成染色体或整个基因组的基因组最小化的探索将大大扩展我们对真核生物核心功能的认识。
4.DNA数据存储技术:BT-IT融合的范例
4.1.新兴的DNA数据存储
信息存储一直是人类文明的推动力,是知识积累、文化传承和技术代代相传的必要条件。用于保存信息的技术可以追溯到古代中国造纸和结绳,以及后来历史上的纸张和印刷。直到大约半个世纪前,基于磁光硅的存储技术,如硬盘、固态硬盘和磁带,不断改变了信息存储的方式。现代数据存储和处理技术使人类进入了数字时代,地球上的数字数据总量呈指数级增长。然而,当前的存储介质正面临一些挑战:密度的理论极限、持续时间短暂、高能耗和环境污染。因此,需要开发新一代信息归档技术。令人惊讶的是,DNA作为保存遗传信息的天然介质,已被发现是潜在的人工数据存储介质,具有高密度、长期耐用性和低维护成本。利用合成DNA进行高密度和长期数据存储已成为一个非常有前景的研究领域,吸引了全球政府和产业投资者的极大兴趣。半导体行业协会、国防高级研究计划局、国家科学基金会、半导体研究公司和情报高级研究计划活动都为美国DNA数据存储技术和相关半导体做出了贡献。欧盟委员会还特别资助了DNA数据存储并启动了地平线2020计划。中国科协在2018年将DNA数据存储列为60个重大科技工程问题之一。中国的第十四个五年计划明确促进了DNA存储等前沿技术的发展。微软和西部数据,以及Twist Biosciences和Illumina在2020年宣布创建“DNA数据存储联盟”。它的“共同目标是发挥DNA数据存储作为现有和新兴归档存储用例中新存储介质的全部潜力”。迄今为止,已有50多家公司和学术机构加入了该联盟。
4.2.DNA数据存储的概念与工作模式
如图3A所示,DNA数据存储的基本概念由三个基本组成部分构成:(i)一种编码系统,能够将二进制字符串编码为DNA字符串,并适应相反的过程——将DNA字符串解码为二进制字符串;(ii)一种写入装置,能够制造具有特定序列或结构的实际DNA分子;(iii)一种阅读装置,能够读取DNA分子的序列。值得一提的是,到目前为止,DNA中数字信息存储有两种不同的策略。对于第一种策略,具有特定序列的DNA分子被用来记录信息。通过从头合成或组装DNA分子来实现数据写入。第二种策略使用预先存在的DNA分子作为数据记录的骨干。然后,通过基因编辑或DNA杂交在双链DNA(dsDNA)或单链DNA骨干上的预定位置存储信息,以在骨干上产生精确的序列或结构修饰。第一种策略提供了更高的存储密度,但由于需要DNA合成,写入成本相当高。第二种策略预计比第一种写入成本更低,因为它绕过了昂贵的DNA合成阶段。然而,其存储密度的降低可能限制了未来的应用。因此,根据写入、复制、存储和读取的技术细节,DNA数据存储技术可以分为体外“硬盘模式”和体内“CD-ROM模式”(图3B和C)。
图3B展示了体外“硬盘模式”,它使用高通量DNA合成来写入数据,并具有高密度数据存储的潜力,类似于普通的硬盘。这种模式下的数据写入和读取过程相对简单,因为没有细胞膜屏障。然而,根据研究,体外存储与DNA链在复制过程中的丢失、高复制成本以及整个存储过程中的DNA降解有关。图3C中的体内“CD-ROM模式”使用人工染色体来存储和分发大量数据。这种体内模型具有一个保护性环境,在其中自然出现了高效的复制和修复酶系统,这在耐用性、保真度和低复制成本方面具有显著优势。与体外“硬盘模式”相比,“CD-ROM模式”的主要优势是染色体DNA作为细胞复制的一部分,其低成本、可靠的复制可以用来快速、低成本的数据复制和传播。此外,体内存储通过构建复杂的细胞内生物电路,如通过基因编辑进行随机读写、加密和解密,以及与生物信息流的通信,更容易实现更高级的存储功能。这些额外的选项为体内模式开辟了更多可能性,允许更广泛的潜在应用场景,如细胞事件记录、环境毒素检测和疾病标记监测。
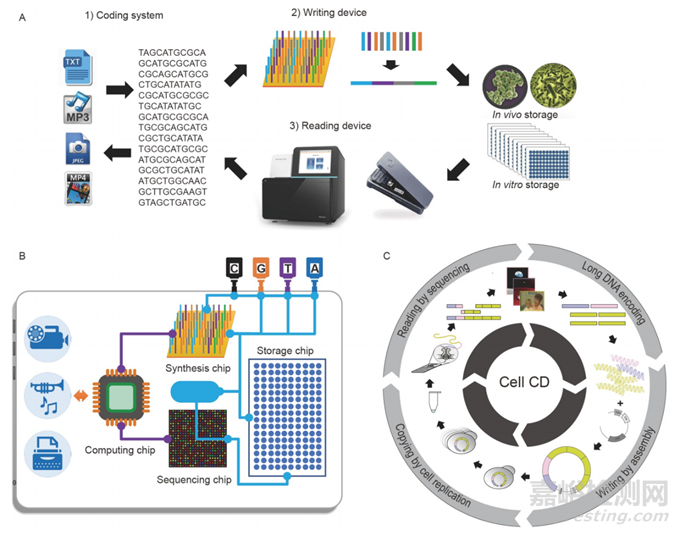
图3 DNA数据存储的基本概念和存储模式。A, DNA数据存储的基本概念。为实现基本的数据写入和读取操作,DNA数据存储需要编码系统、写入设备和读取设备这三个基本组成部分。B, 基于体外合成和测序大量DNA片段的“硬盘模式”。C, 基于体内染色体DNA操作的“CD-ROM模式”(参考Chen等人,2021b的图重新绘制)。两种存储模式的详细信息在正文中有描述。
4.3.DNA数据存储的主要进展和现状
体外“硬盘模式”的可行性已在实验室规模上得到证明。哥伦比亚大学的研究人员引入了喷泉码到DNA数据存储中,以提高编码效率,防止GC富集、复杂的DNA序列难以构建和测序。在2 MB(兆字节,10^6字节)的数据规模下,实现了高达215 PB/g DNA(PB,拍字节,1×10^15字节)的高存储密度。2018年底,华盛顿大学的研究人员在200 MB(200兆字节)的规模上实现了可靠的随机数据访问。此外,构建了一个完全集成的DNA存储系统,实现了单个单词“hello”的自动写入、存储和读取。初创公司CATALOG采取了不同的方法,使用“DNA活字”进行高速数据写入。他们在2019年宣布,可以在12小时内将所有16 GB(千兆字节,10^9字节)的维基百科文本写入DNA,这比其他任何当前使用的技术快近1000倍。以色列理工学院的研究人员设计了一种复合DNA字母的概念,通过利用碱基组成信息来提高每个合成周期的写入能力,从而最小化写入成本。Gao等人使用等温扩增完成了低偏差DNA链扩增。天津大学的Chen等人使用低密度奇偶校验(LDPC)和里德-所罗门(RS)算法对总计3 MB的视频片段和文本进行编码。为了解决DNA存储的序列兼容性问题,Ping等人设计了一个“阴阳”编码系统。早期的大肠杆菌DNA CD-ROM范例概念研究使用了质粒存储数据。后来的研究集中在实现遗传电路,如切换开关,用于数据存储。然而,这类系统的存储容量显然有限。Shipman等人利用CRISPR-Cas9技术在细菌细胞中存储了数字电影,并使用高通量测序使它们能够被解码。后来,Tang和Liu使用两个CRISPR介导的模拟多事件记录装置系统记录了大量细胞活动。
最近,天津大学的Chen等人从头设计并合成了一个长度为254,886 bp的人工染色体用于数据存储。这项研究表明,首次组装的人工染色体可以通过可靠和低成本的细胞复制用于大规模数据分发。“万物DNA”、“生物正交信息存储”、“真实随机数生成”、“DNA中的数据加密”等新概念和想法也已被提出,为DNA存储和计算的广泛潜在应用铺平了道路。最近的一项综述为这些主题提供了极好的总结。
4.4.未来展望
DNA数据存储涉及一系列关键技术,包括DNA合成、测序、微流控技术、微纳米制造等,需要跨学科的努力,以实现将DNA存储转化为实际应用的最终目标。尽管之前的研究在数据量、稳定性和随机访问方面取得了显著进展,但成本,尤其是写入成本,已成为DNA数据存储实际应用的主要障碍。据估计,与当前使用的基于磁带的存储技术相比,DNA数据存储需要将写入成本降低7-8个数量级。尽管有几次尝试,例如未终止的TdT、DNA打孔卡、DNA活字、复合DNA字母和低质量合成,但成本降低的竞争途径仍然不明确。每种信息存储介质在其早期阶段都面临着相同的高生产成本挑战。
现代存储技术已根据摩尔定律广泛使用了几十年。值得一提的是,DNA合成和测序,作为DNA数据存储的两项关键技术,其发展速度超过了摩尔定律的预测。在DNA存储的简短历史中,自从Church等人在2012年首次发表基于芯片的DNA数据存储以来,数据量已经扩大了300多倍,显示出快速上升的趋势。
总之,作者认为,随着酶促DNA合成、数据写入和读取方法的不断发展,实用的DNA数据存储技术将在未来几年内实现。作为一种环保、高容量、长期存储介质,DNA预计将弥补当前存储介质的不足。
5.基因编辑
在生命科学领域,长期以来一直有一个目标,即能够以编程方式、特异性和高效地编辑所有活细胞的DNA序列,这在基因研究、基因治疗、遗传育种和合成生物学中具有无限的价值。以前的方法,如巨型核酸酶、锌指核酸酶(ZFNs)和转录激活因子效应物核酸酶(TALENs),依赖于复杂和特定的蛋白质-DNA相互作用,将蛋白质效应子定位到所需的DNA序列。虽然这些方法在针对特定位点方面是有效的,但要快速而简单地重新编程这些蛋白质结构域以针对新的基因组位点是困难的。成簇的规律间隔短回文重复序列(CRISPR)系统的发现和工程引发了基因组编辑领域的新而激动人心的复兴。
5.1.严格基于蛋白质的基因组编辑系统
巨型核酸酶、ZFNs和TALENs是强大的生物学工具,可用于基因组编辑(图4A)。
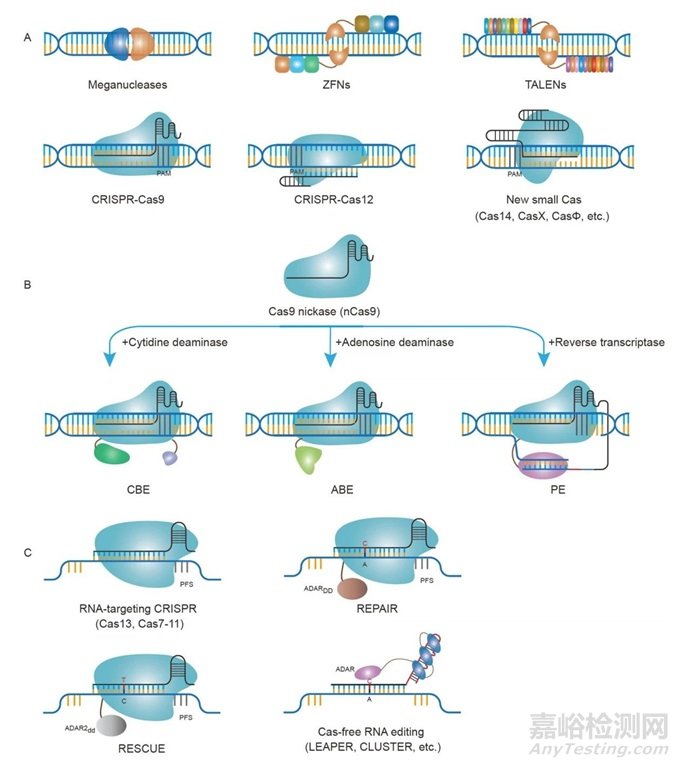
图4 基因组编辑技术的概览。A, 基于核酸酶的基因组编辑技术,针对DNA,包括巨型核酸酶、ZFNs(锌指核酸酶)、TALENs(转录激活因子效应子核酸酶)、CRISPR-Cas9、CRISPR-Cas12以及新的小型Cas变体。B, 精确DNA基因组编辑技术,包括胞嘧啶碱基编辑器、腺嘌呤碱基编辑器和首要编辑器。C, RNA编辑技术,包括CRISPR-Cas13、CRISPR-Cas7-11,以及像REPAIR、RESCUE等RNA碱基编辑方法和其他无Cas的RNA编辑方法。
巨型核酸酶(也称为归巢内切核酸酶)是大型蛋白质复合物,可识别特定的DNA序列。这些蛋白质依赖于蛋白质本身与目标DNA序列之间的复杂相互作用网络。尽管以前的努力已成功地将巨型核酸酶应用于新的、用户定义的基因组序列,但这一过程极其繁琐、耗时且技术上具有挑战性。巨型核酸酶的应用从根本上依赖于大规模重新编程整个蛋白质复合物,使其能够识别感兴趣的新DNA区域。因此,迫切需要使用需要变体库的高通量方法来识别针对新定义的蛋白质序列的新变体。因此,需要更可编程的方法用于热稳定的DNA靶向。
锌指蛋白是能够识别特定三个DNA碱基序列的小蛋白质模块。这些蛋白质在自然界中很常见,以前的研究已经确定了决定特定3个碱基对DNA结合序列的单个锌指模块的关键组成部分。可以将模块化锌指阵列融合在一起,以实现基于特定DNA序列的DNA靶向。此外,研究人员巧妙地将这些参与DNA靶向的较大的锌指蛋白与FokI蛋白融合,FokI蛋白可以切割DNA。为了最小化活细胞中所有不需要的随机DNA切割,研究人员巧妙地将FokI蛋白分成两半,每半都使用特定的锌指招募到DNA的目标区域。因此,两个锌指核酸酶(ZFNs)的结合可以特异性和精确地切割DNA。这些ZFNs已在人类、动物和植物细胞中显示出功能,因此在可编程基因组编辑中发挥重要作用。
在发现锌指蛋白之后,研究人员从植物病原体中鉴定出转录激活因子样(TAL)效应物。与ZFs不同,每个TAL效应物(TALE)结合一个DNA碱基。这种效应可以被编程以结合特定的DNA序列。TALE与FokI二聚体结合,生成TALE核酸酶(TALENs),这是一种完全基于蛋白质的可编程基因组编辑技术。与ZFNs相比,TALENs表现出更好的编程能力,因为每个DNA碱基由一个单独的单元识别,而不是锌指的三联体编码特性,但TALENs比ZFNs更大,因此传递仍然是具有挑战性的。此外,需要构建蛋白质复合物,当寻求广泛编辑活细胞的基因组时,这并不容易。
5.2.CRISPR-Cas系统
在研究细菌基因组时,研究人员发现了一种名为成簇规律间隔短回文重复序列(CRISPR)阵列的重复DNA序列。通过随后的研究,研究人员证明CRISPR阵列及其附近的蛋白质,CRISPR相关(Cas)蛋白,作为细菌针对外来入侵核酸的免疫系统。当细菌暴露于病原体DNA片段时,免疫系统分离一小部分外来DNA,并将其序列整合到细菌基因组中的CRISPR阵列本身。这一发现对于CRISPR-Cas作为革命性基因组编辑技术的发展至关重要。
CRISPR阵列被鉴定为编码与Cas蛋白相关的RNA序列,并针对DNA中的基于间隔序列的核酸序列。随后的工程证明,CRISPR RNA中的靶向序列可以被轻易替换并用用户定义的序列编程(图4A),这将完全改变并重新编程Cas蛋白的靶向序列。这一发现在基因组编辑领域发挥了重要作用,因为这是第一次基因组编辑试剂可以通过替换核酸序列轻易重新编程,不同于以往需要复杂和高通量蛋白质工程的方法。一旦与目标DNA序列结合,Cas蛋白就会启动双链DNA的切割,从而在活细胞的基因组中造成损伤。
5.3.新的CRISPR蛋白
Cas蛋白是CRISPR基因组编辑技术的关键组成部分。化脓链球菌(Sp)Cas9是第一个用于基因组编辑应用的工程化Cas蛋白,并且在开发新的编辑技术时仍将被广泛使用。所有Cas蛋白已知需要一个原间隔子相邻基序(PAM),这是一小块直接位于目标基因组位点旁边的DNA。Cas蛋白的这种靶向范围限制在尝试在细胞基因组的其他位置进行编辑时仍然是一个挑战。研究人员已经发现了大量具有不同PAM要求的新Cas蛋白,从而扩大了CRISPR基因组编辑技术的靶向范围。此外,蛋白质工程和定向进化的努力已成功改变了Cas蛋白的PAM要求,这有助于利用CRISPR Cas开发一系列基因组目标。
最近,许多新的小型Cas蛋白被发现。SpCas9的长度为1368个氨基酸,这在碱基编辑器和引导编辑器中通过效应蛋白进一步延长。基因组编辑蛋白的稳定性和传递受到增加长度的负面影响。新的CRISPR-Cas蛋白,如Cas12f,以前称为Cas14,CasΦ,CasX,比以前发现的许多Cas蛋白更小(图4A)。然而,需要进一步的工程、发现和进化努力来提高这些新Cas蛋白的编辑效率。
5.4.遗传敲除
巨型核酸酶、ZFNs、TALENs和DNA靶向CRISPR-Cas系统都通过切割双链DNA来运作。在产生DNA双链断裂(DSBs)后,细胞的内源性修复机制迅速修复这些损伤。完美的修复可以作为额外编辑的基质,直到非同源末端连接(NHEJ)或微同源介导的末端连接(MMEJ)修复在目标位点周围产生随机小的DNA插入或删除(INDELs)。INDEL导致基因敲除,这在某些情况下很有用,但缺乏精确性。同源定向修复(HDR)是一种竞争性修复过程,其中使用核酸供体模板来修复DNA。尽管可编程,但HDR与NHEJ/MMEJ修复相比极其低效。因此,需要新的基因组编辑技术来高效、精确地编辑DNA序列。
5.5.碱基编辑
碱基编辑是一种可编程、高效且精确的基因组编辑技术,它建立在将DNA结合蛋白定位到感兴趣序列的能力上。设计的第一类碱基编辑器称为胞嘧啶碱基编辑器(CBEs),利用Cas蛋白结合并解开目标区域的DNA成单链DNA状态的能力(图4B)。CBEs由一个单链特异性胞嘧啶脱氨酶组成,该酶与Cas蛋白融合,通过Cas蛋白靶向的内源性DNA区域进行脱氨。DNA中胞嘧啶碱基的脱氨会产生尿嘧啶,尿嘧啶可以通过复制和修复被细胞内过程转化为胸腺嘧啶。为了提高编辑效率,CBE还包括尿嘧啶糖苷酶抑制剂(UGI),以抑制细胞内尿嘧啶N-糖苷酶(UNG),该酶特异性识别细胞基因组中尿嘧啶碱基的存在。局部UGI的存在将延长尿嘧啶中间体的寿命,从而促进修复后胸腺嘧啶的永久性整合。
为了进一步促进编辑,Cas蛋白被转化为一种切口酶,它切割与被编辑链相对的链,使用含有新碱基编辑尿嘧啶的DNA的对侧链作为修复模板,操纵细胞修复机制来替换被切割的痕迹链。这最终实现了从一条DNA链到两条DNA链的永久性编辑,显著提高了碱基编辑的效率。
腺嘌呤碱基编辑器(ABE)是开发的第二类碱基编辑器。ABE由一种实验室进化的腺苷脱氨酶组成,该酶将DNA中的腺嘌呤碱基转换为肌苷(图4B)。肌苷随后被细胞内聚合酶识别为鸟嘌呤。先进的定向进化方法进一步提高了编辑效率,并扩展了腺嘌呤碱基编辑的用途。
最初的碱基编辑方法使用Cas蛋白解开DNA,暴露单链DNA序列作为脱氨的底物。一类新的碱基编辑器,称为DddA衍生的胞嘧啶碱基编辑器(DdCBEs),利用一种自然存在的双链DNA胞嘧啶脱氨酶DddA,在不解开DNA的情况下执行碱基编辑。DNA结合蛋白,如TALE或ZF,可以与分裂的DddAs和UGIs融合,指导胞嘧啶碱基编辑到目标DNA序列,而不需要Cas蛋白。此外,通过将催化受损的DddA变体与腺嘌呤脱氨酶TadA8e融合,在人类线粒体中实现了目标A到G的编辑。
CBE、ABE和DdCBE都可以精确高效地编辑DNA,以创建CG到TA(CBE和DdCBE)或AT到GC碱基(ABE)的转换。然而,还有许多其他类型的基因组编辑,例如其他碱基转换和可编程的插入和删除,这些需要更新的精确编辑技术。
5.6.引导编辑
引导编辑是一种精确的基因组编辑技术,它使用Cas蛋白结合DNA并切割DNA的一个链的能力(图4B)。与碱基编辑器不同,引导编辑器在Cas结合后切割解开的单链R环DNA。在这个链被特定切割后,释放的DNA可以作为引物执行随后的DNA聚合。
引导编辑的另一个关键组成部分是引导编辑引导RNA(pegRNA),它编码一个与Cas蛋白切口释放的单链R环互补的引物结合位点,以及编码特定所需DNA编辑事件的模板区域。在RNA-DNA杂交之后,与Cas蛋白融合的逆转录酶蛋白可以使用pegRNA作为模板来扩展基因组DNA。一个针对引导编辑3'区域的正交Cas蛋白引导RNA可以进一步增强编辑。在随后的DNA复制和修复之后,新合成的DNA序列可以永久地整合到基因组中,从而实现由pegRNA序列决定的可编程和多功能的编辑。引导编辑最初的演示编辑效率相对较低,然而,程序的后续修改,如最佳引物结合熔解温度,使用两个pegRNAs,DNA修复操作,RNA稳定性基序和逆转录酶酶的修改已经大大改善了引导编辑效率。
5.7.RNA编辑
RNA中的基因组编辑可以避免对基因组的永久性变化,从而降低非目标DNA编辑的风险。一类针对Cas蛋白的RNA,如Cas13a和Cas7-11,可编程地靶向由CRISPR引导序列确定的RNA序列(图4C)。类似于DNA碱基编辑器的发展,研究人员通过将RNA特异性腺苷脱氨酶与靶向RNA的Cas蛋白融合,开发了RNA碱基编辑器(图4C)。RNA特异性腺苷脱氨酶(ADAR)蛋白通过“RNA编辑与可编程的A到I替换(修复)”技术与Cas13a融合,将腺嘌呤转换为肌苷(与ABE DNA碱基类似)。同样,能够脱氨RNA中的胞嘧啶的工程化ADAR蛋白被用来开发“特定CU交换的RNA编辑(RESCUE)”技术,该技术将RNA中的胞嘧啶碱基转换为尿嘧啶(与CBE DNA碱基编辑器类似)。
已经开发了新的不依赖CRISPR的RNA编辑系统,通过利用RNA核酸招募内源性蛋白质与RNA发生化学反应的能力,执行位点特异性的RNA编辑(图4C)。此外,一种并行技术表明,较长的RNA可以自然地招募ADARs进行RNA的A到I编辑。最近,RNA的聚集设计和环设计显著提高了RNA编辑技术的编辑效率和特异性。
过去十年标志着新基因组编辑技术的快速发展。从最初的基于蛋白质的方法到如引导编辑这样的精确基因组编辑技术,操纵活细胞和生物体的基因组的能力越来越令人兴奋。RNA编辑技术也开始变得更精确和高效。迫切需要继续发展更小、更精确、准确和高效的基因组编辑工具,特别是在治疗、农业和生物研究等领域的应用。
5.8.基因组编辑的应用
基因组编辑技术的发展在生物医学和农业领域取得了巨大进步。生物技术公司纷纷推进新的基因组编辑药物的研发。最近,研究人员在体内基因组编辑技术方面取得了进展,如CRISPR-Cas9和碱基编辑,用于治疗镰状细胞性贫血、早衰症、转甲状腺素淀粉样变性或高胆固醇血症等遗传疾病。基因组编辑在农业中的应用激发了对未来生物作物育种的新兴趣。抗病性和除草剂抗性是许多作物种类中发展最为成熟的两个方面。最近,研究人员展示了通过四次同时多路编辑事件,成功创造了抗病性和产量增加的小麦植物。此外,许多内源性编辑已被证明可以产生强大的除草剂抗性。基因组编辑将继续促进有价值的农业作物品种的创造。
6.蛋白质的分子进化
体外蛋白质分子进化加速了蛋白质的自然进化过程,为蛋白质科学和应用创造了无限机会。该方法的最初贡献者Frances H. Arnold获得了2018年的诺贝尔化学奖。近年来,人们致力于构建更有效的定向进化方法,这不仅有助于深入理解蛋白质的基础科学,还可以创造优于自然或不存在的酶和抗体,并促进合成生物学的应用。
6.1.基于结构的进化
一种基于对蛋白质结构-功能关系的深刻理解的策略,称为合理设计,可以在短时间内产生所需的突变体。精确定义突变“热点”是实现期望结果的关键。此外,构建更小但更智能的突变库可以显著加快进化过程。
随着生物信息学的快速发展,“热点”预测变得流行,因为某些残基位置上的突变限制的释放可能对酶的特定功能产生重大影响。已经开发出计算工具来识别和评估有利的热点。例如,ConSurf网络服务器可以分析蛋白质结构的进化保守模式,LigPlot+程序可以生成蛋白质-配体相互作用的示意图,CAVER 3.0可以可视化隧道和通道中的蛋白质结构。PoPMuSiC网络服务器可以估计最近的蛋白质稳定性变化,算法ASRA和Innov’SAR非常适合作为在结合口袋内饱和突变的指导,以增强立体选择性和活性。
随后开发了各种专注于活性位点工程的稳健策略,并已用于脂肪酶、葡聚糖酶、木聚糖酶和其他酶进化的主要成就。通过结构和系统发育分析,环重塑在几个突变步骤内重新构建了具有PTE样乳糖活性的磷酸三酯酶(PTE),展示了环重塑在快速区分新酶功能方面的潜在作用。逐步环插入策略(StLois)通过结构和功能分析相应的酶,有效地扩展了环区域的残基,为新催化属性提供了新的酶活性位点结构。结构域交换有助于揭示重要调节器的结构和功能信息,例如β抑制因子和衰减加速器。
半合理设计在选定的残基上引入随机突变,饱和突变在选定的残基上创建了一个包含所有可能突变的小突变体库,其中一些可能对突变蛋白有益。值得注意的是,在密码子简并性的帮助下,组合活性位点饱和测定(CAST)和迭代饱和突变(ISM)的有效构建。在创建“智能库”方面取得了显著进展。这些方法已被报道成功改善了酶的性质,如热稳定性、催化活性和对映体选择性。酶工程与系统代谢工程的结合也显著增加了目标产物的代谢通量。
6.2.随机突变
定向进化不依赖于酶的结构信息,而依赖于酶的序列信息,为实验室在几个月内而不是数百万年获得所需的突变体提供了有希望的方法。变异的序列空间非常大,例如,在四个残基上突变可能会产生160,000(20^4)个序列。定向进化中的一个关键问题是如何有效地生成突变体库。通用方法是错误倾向PCR(epPCR),它引入了基因的变化。研究人员通过改变PCR反应条件显著增加了突变率。Zaccolo等人通过改变PCR条件和突变PCR循环次数,将突变率重新调整为每五个碱基对一个突变。到目前为止,epPCR已经取得了许多成功,例如提高酶和底物的活性、亲和力和稳定性。最重要的是,epPCR也是通过分析大规模序列多样性来研究分子进化的强大方法。
DNA重组模仿自然同源重组,这是自然进化的另一种机制。在DNA重组期间,两个或多个相关的起始基因被重组,产生具有新组合的随机序列的变异基因池。与epPCR相比,DNA重组结合了相关功能蛋白的片段,产生新序列,这些序列与所需的蛋白结构和功能兼容的可能性相对较高。一个例子是从过氧化氢酶生成具有改变的选择性的活性卤素酶变体,以扩大未激活的C-H键的酶促卤化能力。同样,基于BRC重复模块性的基序重组被用来生成更强的嵌合体,与RAD51结合。
最近,已经开发了许多有前景的技术用于体内蛋白质进化(图5)。这些方法通过将突变酶或核酸酶定位到DNA中,直接在宿主生物体内生成多个随机突变。CRISPR/Cas9开创了基因组编辑的新时代,也已应用于蛋白质工程。EvolvR系统由Cas9-nickase和连续在gRNAs的指导下产生可调窗口中突变的错误倾向DNAP I组成。更具体地说,一种新的体内突变方法,CRISPR-Enabled Traceable Genome Engineering(CREATE),利用CRISPR/Cas9系统和条形码跟踪盒突变多个位点并跟踪它们。它可以为整个蛋白质序列形成单碱基库,构建一个饱和库,其中每个氨基酸残基都被替换。噬菌体辅助顺序进化(PACE)是另一种体内进化策略。它利用M13噬菌体在大肠杆菌中突变基因的生存能力。一般来说,PACE可以进化任何与基本噬菌体基因表达相关的蛋白质。由于噬菌体生命周期的快速代时间,一天内可以在没有人为干预的情况下进行数十轮进化。此外,由于T7 RNA聚合酶对DNA的结合亲和力,它被用于几个体内蛋白质进化系统中。MutaT7是一种包含T7 RNA聚合酶和胞嘧啶脱氨酶的嵌合蛋白,可以编辑或突变T7启动子下游的特定基因。最近,基于T7 RNA聚合酶的定向体内多样化(TRIDENT)已经开发出来,它基于T7 RNA聚合酶的进化平台,利用增加的突变多样性和更高的体内突变率。
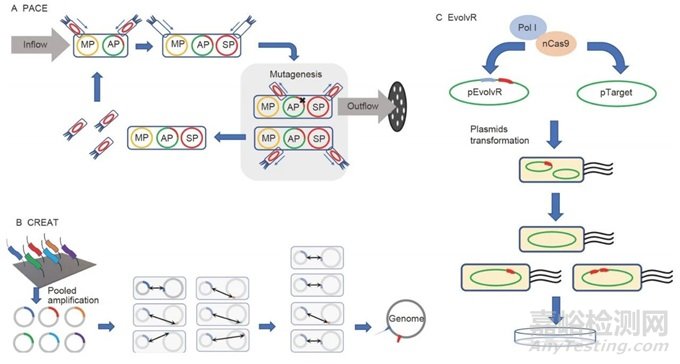
图5 正在兴起的体内突变方法概览。A, PACE的原理图。PACE利用M13噬菌体的存活来在大肠杆菌中突变基因。MP,诱变质粒;AP,辅助质粒;SP,选择质粒。B, EvolvR的原理图。EvolvR利用一个由错误倾向的Pol I和Cas9的切口变异体组成的嵌合蛋白,通过gRNA靶向特异性突变基因。nCas9,Cas9的切口变异体。C, CREATE的原理图。CREATE利用带有条形码追踪突变的CRISPR-Cas9进行多重基因组工程。
6.3.高通量筛选
高通量筛选是一种从大型变体库中获得所需突变体的技术。基于微孔板的筛选方法是酶定向进化中最常用的方法。这些系统具有易于安装、操作方便和强大的多功能性的优点。然而,筛选能力相对较低,通常限于每天103-104个菌落。为了加快筛选速度,已经开发了自动化设备,如机器人液体处理单元和菌落挑选系统。在高通量筛选方法中,通常使用比色或荧光底物来测量酶活性。这种筛选还可以与pH指示剂或产生吸收或荧光信号的酶级联反应结合,以创建高通量筛选方法。
生长互补选择是一种强大的筛选方法,每当目标酶对宿主细胞生存至关重要时。这种方法已广泛应用于与主要代谢途径相关的酶,包括tRNA合成酶、蛋白酶、氨基酸合成异构酶等。同样,可以通过拯救含有关键位置点突变的缺陷抗生素抗性基因来筛选具有所需功能的酶,例如碱基编辑器。
荧光激活细胞分选(FACS)和荧光激活液滴分选(FADS)具有大于10^6个/小时的筛选通量,使它们成为超高通量筛选技术基准。在开创性研究中,为糖基转移酶设计了可以在细胞内外自由移动的荧光底物,荧光产物可以在细胞内捕获并通过流式细胞仪筛选。对于不能吸收所需底物或保留荧光信号的细胞,FADS使用液滴作为酶微反应器来分离单个细胞。微流控芯片系统允许进行多种操作,如液滴产生、细胞裂解、试剂添加、孵育、荧光检测和双通道筛选。最近,发明了一种结合FACS和FADS的方法,其中可以使用商业FACS仪器选择完整的双乳液滴。
蛋白质展示技术是筛选蛋白质或肽结合活性的重要平台。噬菌体表面展示首次用于研究抗原-抗体结合。随后发明了各种细胞展示方法,如细菌展示和酵母表面展示。细胞展示方法也广泛用于定向进化,如提高β-内酰胺酶的稳定性和扩大DNA聚合酶的底物谱。同样,无细胞展示方法,如核糖体展示和mRNA展示,加速了酶的定向进化。
与噬菌体展示相比,细胞表面展示提供了更大的展示表面,并且如果相关荧光检测可用,也可以通过FACS/FADS进行筛选。此外,无细胞展示系统克服了基于细胞的展示方法对转化效率的限制,因为它可以处理高达10^14个成员的库,并且也适用于生成有毒或不稳定的蛋白质。
7.计算机辅助设计功能蛋白
蛋白质是具有丰富生物功能的细胞大分子,构成生物系统的基本构建块。然而,由于蛋白质系统的序列结构-功能空间相当大,数学解决蛋白质相关问题极其具有挑战性。蛋白质的有效设计,合成生物学的核心任务之一,以可接受的准确性为代价显著压缩了搜索空间。计算蛋白质设计的目标是使用算法创建能够折叠成特定结构并具有所需功能的蛋白质。随着蛋白质结构计算预测的突破和序列设计算法的不断出现,已经有可能开发支持合成生物学的计算蛋白质设计平台。
7.1.设计蛋白质结构的算法
目前,蛋白质序列通常根据现有蛋白质结构的数据,按照固定的主链进行设计。与给定的狭窄结构空间相比,相应的蛋白质序列空间相当大,表型负影响可以显著削弱设计蛋白质的可折叠性。因此,序列设计需要开发针对性的算法和策略。计算蛋白质设计常用的方法可分为以下几类。(i)主链生成:根据序列设计的要求构建主链构象模型。(ii)侧链布局:根据给定的蛋白质框架结构,选择一组合适的氨基酸侧链构象以满足主链结构的要求。这需要实际设计序列,也称为蛋白质序列设计。(iii)刚体放置:固定蛋白质/蛋白质或蛋白质/小分子之间的相对空间位置和方向。(iv)负膜设计:增加非目标状态的能量并实现有效折叠,可以视为侧链布局算法的优化和补充。
计算蛋白质设计通常涉及三个步骤。首先,在主链上放置离散的侧链构象。接下来,计算插入的侧链与原始侧链之间的能量以及侧链与主链之间的能量。最后,通过搜索算法优化序列和构象的组合(图6)。整个过程涉及通过搜索算法优化一系列序列组合及其相应的结构。预先给出固定的主链框架(例如,派生于天然蛋白质结构)。每个主链位置上的氨基酸残基类型及其侧链构象未知,应进行计算。不同位置的残基选择和结构状态的可能组合构成氨基酸序列和侧链结构空间。在此空间中定义的能量函数用于评估特定的序列和构象组合。搜索算法自动搜索未知数量的序列空间和侧链构象,以找到设计蛋白质结构的最低能量解决方案。为了正确模拟突变的侧链构象,需要重新设计现有结构。这一步通常使用软件的依赖于主链的转子分子库执行,而侧链的优化是能量依赖的。
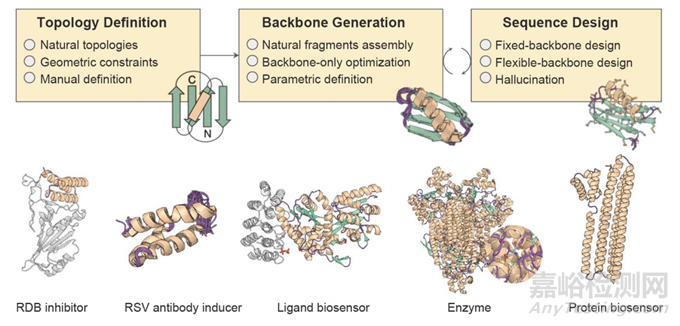
图6 计算机辅助蛋白质设计的原理和实例。
能量函数是表征每个序列组合不同构象结构的基础。不同的算法使用不同的能量函数,主要包括物理能量项(非共价范德华相互作用、静电能量、氢键能量、溶解自由能)和统计能量项(主链二面角、侧链扭转)。最广泛使用的能量函数是Rosetta能量函数(主要由物理能量项决定)和基于主链的氨基酸使用调查(ABACUS)/侧链未知主链排列(SCUBA)能量函数(主要由统计能量项决定)。
在固定主链的蛋白质设计中,共价键的长度和角度通常是恒定的,需要考虑的主要相互作用是非共价的。在Rosetta能量函数中,使用Lennard-Jones势计算范德华相互作用能量。静电能量是使用CHARMM分子力场的原始原子电荷分布计算的,并通过团体优化进行调整。氢键能量是使用静电模型和特殊的氢键模型计算的,氢键被分类为四种类型:长程主链氢键、短程主链氢键和主链与侧链原子之间的氢键。侧链之间的氢键是单独计算的。Lazaridis-Karplus隐式高斯排斥模型可以包含各向同性和各向异性的溶解自由能,以描述溶解效应。统计能量项代表通过将数据库中存在的概率分布转换获得的能量。从统计热力学的角度来看,在平衡状态下,系统不同微观状态的能量和概率遵循玻尔兹曼分布。
另一种观点是,从纯粹的统计角度来看,假设给定主链结构的氨基酸序列分布可以写成条件概率,序列设计要解决的问题是找到具有最大条件概率的序列。因此,ABACUS结合了不同的结构特征:氨基酸位置的结构类型;主链二面角;溶剂可及性;相对位置;以及残基之间的统计信息,以获得侧链转子(转子是旋量异构体)和原子包装能量。此外,SCUBA利用神经网络从以主链为中心的结构变量能量景观中学习显式能量项。SCUBA和ABACUS一起,为人工蛋白质的设计提供了全面的解决方案。
搜索算法对于蛋白质序列设计也至关重要,以避免在相当大的序列空间中遍历所有构象组合,甚至是更大的构象空间。因此,作为一种随机软件,Rosetta基于蒙特卡洛方法设计,以执行多次模拟生成的构象的统计分析,然后获得数值解。Rosetta首先使用随机数生成器生成随机图像。然后确认随机扰动,并评分新构象,接受所有评分更高的构象和以一定概率评分较低的构象,直到在给定的周期内选择最佳评分。然而,这种迭代算法通常陷入局部最小值。为了获得全局能量最小构象,除了分子动力学模拟,还使用了物理概念的动量。想象一个小球从高能函数滚下来。当动量足够高时,球不会被困在小坑里,而是会冲向最后的峡谷。迭代不仅考虑当前能量,还考虑之前的能量变化。
基于统计和机器学习提出了几种算法。受到结构预测算法trRosetta成功的启发,Baker等人进一步开发了Hallucination蛋白质从头设计方法。首先,将随机序列输入trRosetta作为输入,以预测残基接触图。然后,使用蒙特卡洛方法对氨基酸序列空间进行采样,并计算序列之间的KL散度,以获得可折叠的序列和预测结构。Hallucination方法提出了基于卷积神经网络的DeepDream算法,该算法将输入转换为训练数据空间,并产生(注意时态)类似梦境的幻觉。因此,Hallucination方法可以用来快速设计与输入序列相似并符合trRosetta学习到的序列结构关系的蛋白质序列,但与自然序列大相径庭。
7.2.合成生物学中的蛋白质设计
从蛋白质结构设计的序列无法直接满足合成生物学对所需功能蛋白的需求。蛋白质的计算设计主要包括蛋白质自身骨架的设计、蛋白质-大分子相互作用和蛋白质-小分子相互作用的设计。这些相互作用可以被工程化,以优化作为合成生物学组成部分的天然蛋白质的功能,同时创建具有所需功能的生物传感器、生物催化剂和疫苗。
蛋白质框架的设计旨在增强天然蛋白质的稳健性,稳定疫苗表位,并在特定条件下修改蛋白质稳定性。为了开发新型冠状抑制剂,并基于新型冠状病毒S蛋白和人类血管紧张素转换酶2(ACE2)的复合物结构,Baker等人使用与S蛋白受体结合区结合的ACE2的螺旋片段作为起点。尝试通过添加两个额外的螺旋来稳定结构。此外,在微蛋白库中使用蛋白质分子对接和蛋白质界面设计,设计了能够在皮摩尔浓度下抑制2019-nCoV的小蛋白。Correia等人开发了TopoBuilder系统,用于从头设计能够稳定复杂预定义构建块的蛋白质。对于不同的表位,作者列举了合适的二维蛋白质拓扑结构,并使用理想的二级结构构建了三级结构模型。这种方法被用来设计同时呈现三种抗原的蛋白质。结合物理能量项、统计能量项和生物信息学分析,吴等人开发了一种基于单点预测算法和“贪婪”算法融合的蛋白质工程贪婪累积策略(GRAPE策略),通过计算重塑PET塑料水解酶,实现了单点突变,将最终突变的热熔温度提高了31°C。
设计蛋白质-大分子相互作用可以用于合成细胞中的信号转导和调节。Baker等人计算设计的生物传感器可以利用信号传导途径中自然发生的相互作用蛋白。在没有检测目标的情况下,传感器的lucCage蛋白的锁定域与笼子域结合。相比之下,在检测目标存在的情况下,lucCage域的末端区域与检测目标结合,lucCage蛋白打开并与传感器的lucKey蛋白结合,激活荧光素酶发出荧光。同一组还设计了逻辑门来调节蛋白质结合,构建了从头设计的背骨螺旋框架,并构建了氢键网络以优化序列。设计了具有特定异二聚体的多个蛋白质对,使用单体或连接单体作为输入。门控单元通过为特定结合设计的氢键网络编码接受不同的输入。
蛋白质和小分子的相互作用设计可以获得新的酶催化组分、转录因子和小分子传感器。通过设计具有底物选择性的酶,可以为直接用于生物工业催化以及新途径设计生成新的生化反应。在这方面,Kortemme等人筛选了与天然蛋白质结构结合的法尼基焦磷酸(FPP)的四个残基结合模块。然后他们设计了可以与各种框架界面结合并进一步优化的FPP调控的生物传感器。Ranganathan等人使用直接耦合分析提取了多重序列比对(MSA)中隐含序列结构函数空间的统计约束。他们设计了一个与天然酶活性相当的莽草酸转位酶。吴等人使用固定主链设计,结合多个并行短时动力学模拟来补偿固定主链和侧链的不均匀采样。因此,获得了天冬氨酸酶催化的非天然氨基酸的水合反应。
7.3.简短总结
在过去的十年中,在计算创建具有定制活性和特异性的功能蛋白方面取得了令人印象深刻的进展。算法开发的惊人速度不断提高了研究人员操纵蛋白质结构和功能的能力。展望未来,预计有许多关键趋势将加速功能蛋白的发现、设计和应用。通过AI预测蛋白质结构的计算方法的进步提高了生物分子社区的信心,随后的功能设计可能在基于模型和基于数据的方法的结合帮助下,为满足目标反应的需求提供途径。随着蛋白质结构数据库和标准实验数据的不断增长,更先进的计算方法将为解释潜在的催化机制创造进一步的研究机会,最终导致对功能蛋白的结构-功能关系有更清晰的认识。基于计算蛋白质设计的相当大的成功,预计未来将见证为合成生物学生成更有效、定制化的蛋白质。
8.细胞和基因回路工程
无论是使用传统的生物工程还是当前的合成生物学,设计具有有益功能的细胞都面临着相当的挑战。在合成生物学时代,工程细胞的一个标志是强调在系统和定量层面设计和重建非自然的细胞行为,这通常需要多个组件形成具有特定拓扑结构和功能的交互网络。这些可设计的生物网络由大分子组成,如蛋白质、DNA、RNA或每个细胞内的任何遗传部分,称为基因回路。值得指出的是,这样的网络可以在逻辑上超越单细胞层面,换句话说,通过直接或间接的细胞间接触或通信形成互动的多细胞系统,称为细胞回路。
工程化细胞和基因回路面临两个基本挑战:(i) 强调正交性和模块性的可用遗传组件;(ii) 提供可预测电路行为的理论指导的电路模块设计原则的知识。此外,设计过程高度依赖于复杂的计算建模能力,以分析和预测更大电路和参数空间中的电路行为。
因此,合成细胞的计算辅助设计将进一步加强自动化和人工智能在未来细胞工程中的应用,我们将在下一节中讨论。
8.1.合成基因回路和定量细胞行为
基因回路的概念源自电子电路,但由于大量组件的生化或生物物理相互作用以及这些组件之间的非线性连接所产生的巨大复杂性,与电子电路有实质上的不同。与天然细胞中的基因回路类似,合成基因回路包括两种基本类型:(i) 基于蛋白质的信号回路(或蛋白质回路);(ii) 转录基因调控回路(或遗传回路)。然而,这两种类型差异很小,并且协调工作以控制细胞功能。具体来说,蛋白质回路通过膜受体蛋白质(或传感器)在更快的时间尺度(从秒到分钟)上处理环境信号,然后将信号传递到下游基因调控回路,发生的时间尺度更长(从分钟到小时)。
在过去几十年中,对合成回路的广泛研究成功构建了具有集成功能的遗传回路,如逻辑门、带通、振荡、适应和极化。虽然这些研究中的许多仍处于概念验证阶段,但这些合成回路的复杂性和规模的增加显著提高了我们设计和构建更有效、更精确的复杂遗传回路的能力。合成回路的主要发展方向是充分利用计算机辅助设计和自动化。为此,需要进行大量的研究工作,包括充分表征和标准化的遗传组件、经过实验验证的构建和模拟硅电路的算法和软件,以及定制开发的自动化实验设备。
值得注意的是,由于遗传操作的困难和各种蛋白质或核酸工具的限制,哺乳动物细胞中的电路工程发展不足。例如,哺乳动物电路工程中使用的启动子数量通常是个位数。在现有的启动子工具箱中,目标基因的转录强度难以连续调节,这成为电路设计中电路参数条件实验验证的主要障碍。此外,许多可诱导的哺乳动物启动子的基因转录动态范围极低,不利于构建需要低基础但高度可诱导的基因转录的电路。与细菌细胞类似,很难预测不同哺乳动物细胞系中启动子强度和诱导的一致性。
蛋白质工程比启动子工程更具挑战性。蛋白质功能由20种氨基酸组成的三维结构决定,这比由4种核酸组成的一维序列要复杂得多。至于传感器,受体工程已成为建立正交细胞间信号传导的重要领域,其结果是感应给定的细胞外信号,如合成细胞因子和生长因子,或将细胞重定向到特定疾病信号。嵌合抗原受体(CAR)激活的T细胞已成为抗癌治疗的重要例子。哺乳动物细胞中存在许多类型的蛋白质,它们在不同水平上建立信号通路,包括蛋白激酶/磷酸酶、蛋白酶、适配器/支架蛋白、转录因子或表观遗传调控蛋白。从病毒中采用的几种蛋白酶工具已被重新用于控制细胞功能的许多水平。最近的研究还表明,基于这些工程蛋白酶的复杂逻辑功能的蛋白质回路构建主要基于这些工程蛋白酶。最后,从头开始的蛋白质设计正变得越来越强大,特别是作为工程化可编程蛋白质-蛋白质相互作用的工具。值得注意的是,AI算法的最近发展在未来蛋白质工程中将发挥重要作用。毫无疑问,蛋白质工具的发展对哺乳动物合成生物学来说是一个困难但必不可少的任务。
哺乳动物合成生物学面临的另一个挑战是自然进化的“黑箱”所控制的复杂行为。这些复杂行为表现出定量特性,其原理仍然不清楚。这些原则几乎支配着所有重要的细胞过程,包括细胞周期、大小和数量的控制、稳健性和异质性、稳态和生长、细胞分化和死亡等。迄今为止,很少有合成生物学研究能够涵盖这些生命之谜。令人鼓舞的是,合成生物学的自下而上方法已经显示出新途径,以比以往认为的更详细地理解复杂生物系统的构建。一个显著的例子是控制许多基本生物过程(例如,细胞周期、昼夜节律、信号响应、节肢动物发生)的振荡回路。作为下一步,这个振荡回路预计将作为一个“中央处理器”,智能控制工程细胞的功能。
我们预计,哺乳动物细胞工程,连同新的工具和技术,将是合成生物学的下一个关键步骤之一。
8.2.基于细胞间通信的细胞回路
哺乳动物合成生物学的一个新兴领域是工程化多细胞系统。这将形成基于细胞间通信的具有特定电路结构和功能的相互作用。对于细菌细胞来说,一个明确的方向是从多样化的自然环境和与疾病相关的肠道到农业重要土壤中重建微生物群落。至于哺乳动物细胞,它们自然存在于多细胞相互作用的背景下,即使在结构良好的器官中也是如此。因此,多细胞水平的细胞工程代表了合成生物学的另一个主要途径。
自然系统中的细胞间通信以三种方式发生:(i) 发送细胞产生并分泌的蛋白质或小分子扩散,并在接收细胞中激活表面受体蛋白或细胞内传感器;(ii) 信号分子(通常是小的第二信使分子)通过通道蛋白传输到直接接触的邻近接收细胞;(iii) 发送细胞上的膜配体和接收细胞上的膜受体之间的直接相互作用。很可能发送细胞的信号触发接收细胞中的转录事件。无论如何,这些细胞级电路将导致在单细胞层面上不会发生的极其复杂的群体行为。细菌和哺乳动物细胞中的空间组织模式可以通过典型的带通电路或逻辑门形成。最近,合成群体感应电路已成功部署以控制细菌和哺乳动物细胞中的细胞群体大小。
然而,这种细胞电路仍处于早期发展阶段。未来需要克服两大挑战。首先,目前研究中使用的信号分子太少。相比之下,人体内存在数百种细胞因子和生长因子,它们参与了大量细胞类型的多重调节。因此,工程化合成细胞因子或其他因素以构建未来的细胞电路是很有吸引力的。其次,设计用于直接接触通信的正交受体和配体对是困难的。最成功的示例是合成Notch(synNotch)信号,它通过细胞外识别域使任何配体结合,并触发可编程的下游基因转录。几项示范性研究已经将synNotch系统应用于空间有序的多细胞结构。
除了与基因电路类似的技术挑战外,结构如何决定电路功能的基本原理难以理解,特别是考虑到细胞群体时空调节的复杂性不断增加。例如,设计具有精确控制的生物稳定性或多稳定性的电路对于合成细胞分化有多大意义?哪些电路拓扑结构能够在治疗疾病时实现高效信号放大,具有高保真度和稳健性?细胞电路如何控制稳态下的细胞群体大小和类型?我们预计这些问题需要在细胞电路层面对基本设计原则进行定量和全面的考虑。
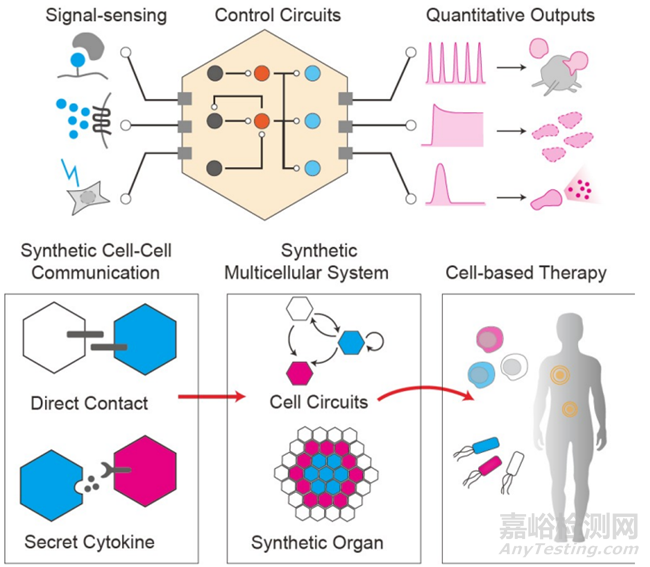
图7 用于治疗的合成细胞和基因回路。在单细胞水平上,工程努力将集中在三个主要方面:(i) 能够识别疾病或环境信号作为生化反应的传感器;然后(ii) 基因回路作为“中央处理器”来处理各种输入信号,产生(iii) 定量定义的输出功能以控制细胞功能。在多细胞水平上,合成细胞因子分泌或直接配体-受体相互作用实现了各种细胞间通信,形成拓扑有序的细胞回路或空间有序的类器官模式。这些单个或多个工程化活细胞可能作为强大的药物平台,用于治疗复杂疾病,如癌症和代谢性疾病。
8.3.工程化活细胞治疗
细胞和遗传电路工程的另一个主要趋势是将目前“玩具”系统中的概念验证扩展到与疾病相关的临床应用。与传统的分子药物形式相比,活细胞药物作为部署有效载荷药物或执行复杂功能的集成平台具有显著优势(例如,细胞溶解、伤口愈合),这些功能可以由集成的基因或细胞电路智能控制。通过这样做,细胞药物可以在最小化副作用的同时显著提高抗病效果。例如,重新配置的细胞因子信号通路可以作为细胞因子开关,感应并消除促肿瘤细胞因子,创造一个促免疫细胞因子的微环境。在CAR-T细胞中,部署具有逻辑门或超敏功能的蛋白质电路,以产生对肿瘤抗原的更特异性识别。CAR-T免疫疗法已经展示了活细胞作为一种药物形式的力量,即细胞疗法。在另一个案例中,通过光遗传学控制的基因电路通过闭环控制策略成功地在动物血液中智能控制了稳态血糖水平。这些引人注目的例子表明,合成活细胞药物已经在治疗难治性疾病方面迎来了新的革命。
由于对人类健康的重要性,目前基于免疫细胞,特别是T细胞的治疗成功主要依赖于使用免疫细胞作为治疗性底盘细胞。最近,其他免疫细胞,如自然杀伤细胞、巨噬细胞,不仅在癌症治疗中,也在治疗传染病方面显示出相当的潜力。虽然许多作为药物工程化的细胞仍处于概念验证阶段,我们预计一旦我们能够设计出更精确和功能性的基因和细胞电路,工程化在原代细胞层面将变得更加容易。一些最近的研究在临床层面上已经显示出了实质性的改进。值得注意的是,多细胞系统将为细胞治疗提供额外的优势,通过将功能性电路模块分配到不同的亚细胞类型中,可以显著降低工程成本。一组具有良好编程的相互作用电路的细胞将作为一个整体工作,实现更有效、更安全、成本更低的治疗功能。
9.无细胞合成生物学
无细胞合成系统代表了与细胞工程平行的合成生物学的另一种技术途径。无细胞合成生物学的目标是一个没有细胞结构的开放系统,专注于所需的代谢网络,使用相应的活性成分,如酶和辅酶,来补充复杂的生化反应。无细胞合成生物学起源于Eduard Buchner关于“非生命酵母裂解物的无细胞乙醇发酵”的范式转变发现。另一个里程碑是Nirenberg和Matthaei对遗传密码及其在蛋白质合成中的功能的发现。对于无细胞合成生物学的发展,已经提出了两种类型的无细胞系统:基于细胞提取物的系统和基于纯化酶的系统。基于细胞提取物的系统一直用于无细胞蛋白质合成(CFPS),以实现细胞外中心法则的基本过程(DNA到RNA,RNA到蛋白质)。基于纯化酶的系统由许多纯化或部分纯化的酶组成,以实现复杂的级联酶反应,主要用于生物制造功能性生物分子和生化产品。与在细胞内进行的系统相比,无细胞合成生物系统具有许多优点,如高产品产量、快速反应速率、高工程灵活性、加速的设计构建测试学习周期、对有毒环境的高耐受性以及易于放大。这些特性使无细胞合成生物学成为许多应用的重要使能技术。
9.1.无细胞生物系统用于蛋白质合成和应用
无细胞生物系统用于蛋白质合成由粗细胞提取物、DNA模板、ATP再生系统、氨基酸、核苷酸、辅因子和缓冲液组成。可以根据要求选择来自大肠杆菌、酿酒酵母、小麦胚芽、兔网织红细胞、昆虫细胞和中国仓鼠卵巢细胞的几种细胞提取物。该系统可用于合成毒性或膜蛋白、生物功能原型设计、蛋白质修饰和生物传感器。
在体内高产量表达毒性蛋白很困难,因为毒性蛋白可能会干扰细胞代谢途径,而膜蛋白总是以包涵体的形式表达。无细胞生物系统可用于合成限制性内切酶、细胞裂解扩张毒素和人类微管结合蛋白等毒性蛋白,因为体外系统对有毒环境具有耐受性。通过添加表面活性剂、脂质体或纳米光盘,可以在无细胞生物系统中表达膜蛋白。许多膜蛋白,如G蛋白偶联受体、四环素泵、ATP合酶和丙型肝炎病毒膜蛋白,都是由基于细胞提取物的无细胞生物系统生产的。
对于生物功能的原型设计,例如遗传组件、遗传电路和代谢途径,无细胞生物系统提供了一个重要的体外平台,并允许在细胞中实现。对于单个遗传组件(启动子、核糖体结合位点和终止子),可以通过PCR突变生成线性表达模板的变体库,然后,在微流控技术的帮助下,可以提取含有单基因变体的细胞,将其封装在皮升液滴中。
除了探测单个遗传组件外,无细胞生物系统还可以用来确定这些组件如何在合成基因控制网络或“电路”中协同工作。已经组装并原型设计了众多无细胞基因电路,包括由正交聚合酶或σ因子顺序表达驱动的级联,以及前馈环和负向自我调节器。为了工程化细胞代谢,无细胞生物系统为阐明这些代谢途径提供了巨大的可能性。使用细胞提取物进行蛋白质合成的无细胞生物系统中,编码酶的DNA模板的表达可以导致途径在单一反应中的自组装,这将是极大的优势。迄今为止,已有几份报告证实了这种方法。例如,分别通过无细胞生物系统从线性表达的DNA中重新识别出包含三个和六个酶的两条途径,分别生产N-乙酰氨基葡萄糖和肽聚糖前体。还将色氨酸转化为purpurin的五酶途径展示出来。此外,最近采用组合策略构建了一个17步的酶途径,用于n-丁醇。结合数据驱动设计,无细胞生物系统可以用来在大肠杆菌提取物中快速评估数百种途径组合,以增强丁醇和3-羟基丁酸在革兰氏阳性厌氧细菌中的生产,展示了无细胞和体内途径性能。
对于包括糖基化、磷酸化、PEGylation和非天然氨基酸(uAAs)的插入在内的广泛蛋白质修饰,无细胞生物系统提供了强大的控制和多功能性,绕过了与基于细胞的毒性和渗透性相关的限制。研究细胞内蛋白质的修饰通常具有挑战性,因为很难在细胞内获得均质修饰的蛋白质。无细胞生物系统已被证明具有高度均质的蛋白质修饰功能。一个经典的例子是蛋白质上特定位点的糖基化。许多治疗蛋白质高度依赖于高效和均质的糖基化。
使用大肠杆菌细胞提取物的无细胞生物系统是检测糖基化的理想的试验场,因为大肠杆菌没有原生的糖基化功能。因此,使用无细胞技术加速无细胞中碳水化合物筛选的能力可能对糖基化治疗和疫苗设计产生变革性影响。开放的无细胞生物系统特别适用于使用由非天然tRNA和氨酰tRNA合成酶组成的正交翻译系统,在mRNA的UAG琥珀色终止密码子上添加uAAs。将uAAs整合到蛋白质中为使用修饰蛋白作为治疗剂提供了无限的可能性。一旦uAAs被整合到目标蛋白质的精确位置,它们就作为生物正交化学手柄,与功能化小分子反应,产生治疗偶联物,如抗体-药物偶联物(ADCs)。
当我们评估无细胞生物系统作为生物传感器的作用时,它们比整个细胞传感器提供了几个实际优势。无细胞生物系统中可以检测到细胞壁不可渗透或细胞毒性的分析物,并且由于整个细胞传感器中可能发生突变和质粒丢失,它们更可靠。无细胞生物系统的蛋白质合成特性可以用来宿主基于基因电路的传感器,这些传感器可以以极高的灵敏度和特异性检测核酸和小分子。为了检测主要来自病原体的病毒和细菌的核酸,将含有病原体的样本中提取的RNA添加到无细胞生物系统中,该系统被编程为仅在存在目标核酸序列的情况下通过设计的toehold开关核糖调节器产生报告蛋白。它可以取代逆转录PCR(RT-PCR)进行更快速的诊断测试。使用这种策略,可以快速检测许多病毒,包括埃博拉、寨卡病毒、诺如病毒、黄瓜花叶病毒、SARS-CoV-2和某些肠道定植细菌。这种无细胞病毒检测系统可以通过冻干技术固定在纸上,以提高其便携性和稳定性,为满足当前Covid-19和未来病毒大流行的紧急诊断需求提供替代方案。
与核酸检测相比,在检测小分子(例如,环境毒素或细胞代谢物)方面,无细胞检测的进展较慢,因为没有合成核糖修饰的类似物用于构建任意小分子的传感器。大多数报道的无细胞小分子传感器检测环境毒素,如汞和氟化物、药物,如γ-羟基丁酸或细菌群体感应信号,如N-丁基-L-高丝氨酸内酯。研究表明,无细胞传感器可以被冻干,即使在纸基质上干燥,也能保持活性数月,为无细胞系统提供了一种替代手段,以满足易于分发和低成本传感的未满足需求。
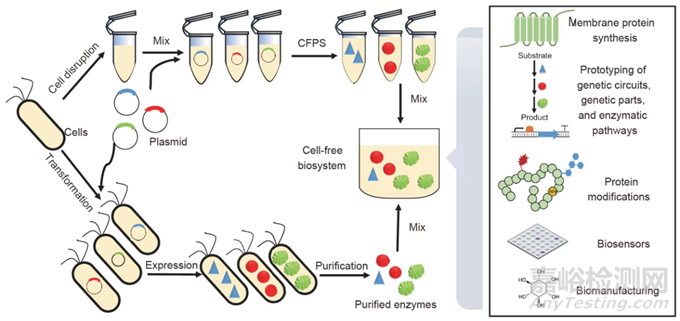
图8 无细胞合成生物学系统的各种应用,包括基于细胞提取物的无细胞蛋白质合成(CFPS)系统和基于纯化酶的系统。
9.2.基于纯化酶的无细胞生物系统用于生物制造
基于纯化酶的无细胞生物系统指的是构建由多个纯化/部分纯化的酶组成的生物催化系统,这些系统通过工程化的反应途径将特定底物转化为所需的化合物。这里,我们专注于使用淀粉、葡萄糖、纤维素和二氧化碳等可持续底物的无细胞生物系统在生物制造中的应用。
肌醇(以下简称肌醇)和氢气是由无细胞生物系统直接从淀粉生产的两个典型产品。肌醇广泛用于化妆品、制药和食品工业。它通过酸水解植酸获得。这种方法使用昂贵的原料,并且产生严重的磷污染。Zhang等人和Atomi等人都构建了一个包含四个酶促反应的无细胞生物系统,可以以100%的理论产率将淀粉转化为肌醇。这个生物系统中的所有酶都是嗜热的,因此可以通过热处理轻松纯化酶,并在高反应温度下避免微生物污染。与传统的化学方法相比,这种从淀粉生产肌醇的新方法具有巨大的绿色生产潜力。
目前,博浩达生物(中国)正在建设一个工业设施,以扩大这种新方法生产肌醇。许多其他附加值化学品,如氨基葡萄糖、阿洛糖和(-)-维博醇,可以通过类似的淀粉酶处理合成。
氢气是未来的交通燃料,通过燃料电池提高能效有潜力减少温室气体排放,为最终用户提供零污染物。自然细胞代谢途径每摩尔葡萄糖只能产生最多4摩尔的H2。Zhang和同事进行了一个概念验证实验,通过包含13种纯化酶的无细胞生物系统,每摩尔葡萄糖产生了12摩尔的H2。该生物系统几乎定量地将淀粉转化为H2和CO2,其总计量化学方程式为:C6H10O5+7H2O=12H2+6CO2。这个生物系统可以稍微修改,以开发能量密度比锂离子电池高一个数量级的糖生物电池。这个无细胞生物系统为氢气生产奠定了基础,为未来的糖-氢车辆。
当葡萄糖被用作无细胞生物系统中的底物时,这里描述的是乙醇、异丁醇和烯烃天然化合物的生产。乙醇是最重要的汽油添加剂,异丁醇是与当前内燃机和运输管道兼容的四碳液体醇。Sieber和同事设计了一个无细胞生物系统,可以通过丙酮酸从葡萄糖生产乙醇和异丁醇。与传统的糖酵解途径中使用的10种酶相比,这个生物系统仅使用四种酶将葡萄糖转化为丙酮酸,然后可以转化为乙醇和异丁醇。即使在4%(v/v)异丁醇存在的情况下,这个无细胞生物系统也能大量生产异丁醇,而即使是低浓度(例如,1%-2% v/v)也会阻止微生物生产异丁醇。这一进展表明,无细胞生物系统对有毒环境具有高度耐受性。为了生产烯烃天然化合物,Bowie和同事设计了一个由20多种酶组成的无细胞生物系统。这些酶可以被划分为4个主要反应模块:一个从葡萄糖生产丙酮酸的糖酵解模块;一个从丙酮酸生产乙酰辅酶A的乙酰辅酶A模块,以及一个从乙酰辅酶A生产萜基焦磷酸(GPP)的甲羟戊酸模块;以及一个生产所需烯烃产物的烯烃化模块。烯烃化模块也可以通过使用替代酶和底物来调节,以生产各种烯烃化合物,如异戊二烯(重复词)和大麻素。经过系统优化后,这个无细胞生物系统产生了1.25 g L−1的大麻素滴度,至少比使用活细胞的结果高出两个数量级。
当纤维素被用作底物时,一个典型的例子是无细胞系统通过纤维素生产淀粉。这个生物系统包含内切纤维素酶、纤维二糖水解酶、纤维二糖磷酸化酶和α-葡萄糖磷酸化酶,用于将预处理的生物质一锅法酶转化生产淀粉。高达30%的纤维素中的脱水葡萄糖单元被转化为淀粉。由于纤维素原料的年来源大约是食品和饲料淀粉的40倍,这种将非食品纤维素转化为淀粉的成本效益转化可以重塑生物经济,并解决食品、生物燃料和环境的三重困境。
中国科学院天津工业生物技术研究所的研究人员构建了一条人工淀粉合成途径(ASAP),利用二氧化碳和氢气合成淀粉。ASAP是一个化学-生物混合系统,包括一个化学系统和一个无细胞生物系统。化学系统将二氧化碳和氢气转化为甲醇。无细胞生物系统包含11个核心酶和3个辅助酶,将甲醇转化为淀粉。在对模块组装、替代和三个速率限制酶的蛋白质工程进行条件优化后,这个化学-生物混合系统以每毫克总催化剂每分钟22纳摩尔的速率将二氧化碳转化为淀粉,这比玉米底物系统中的速率高出8.5倍。这种方法提供了一种潜在的全球粮食供应策略,更重要的是,为探索其他星球时的食物来源问题提供了潜在的解决方案。
总之,无细胞合成生物学提供了一个改变游戏规则的工具,可以绕过活细胞固有的限制。在不同领域,包括基因表达、遗传网络、蛋白质修饰、按需生物传感和使用无细胞生物系统进行生物制造的研究中,无细胞合成生物学的前景是显而易见的。然而,要实现无细胞生物系统的真正潜力,需要克服几个挑战,包括这些生物系统的寿命和不稳定的天然辅因子的再生。在解决这些缺陷之后,无细胞合成生物学将把生物学和生物技术带入一个新时代,带来许多有趣的结果。看到无细胞合成生物学与材料科学、电子学、计算机和人工智能等其他尖端学科相结合,这是令人兴奋的。
10.人工智能和合成生物学
获得理想的生物组件是构建合成生物学系统的基础。随着最近计算能力的增加,人工智能(AI)已被证明在各种具有挑战性的任务中表现出色,例如图像生成、自然语言处理和合成生物学应用。本节将简要描述在挖掘复杂生物学属性和设计优化的合成生物学组件(生物部件)方面越来越成功的AI依赖方法,特别是基因调控序列。有关特定应用的深入讨论,如代谢工程、基因治疗和药物发现,有优秀的综述可供参考。
10.1.AI引导的生物部件逆向设计的一般框架
生物部件设计是一个重要但复杂的任务,旨在基于特定目标属性逆向工程新的生物分子。从实验上讲,穷尽搜索潜在的序列空间以发现新的生物部件(例如,100个碱基对的DNA序列形成一个潜在的序列空间4^100)是非常困难的。因此,虚拟筛选为探索这个广阔的空间提供了一个有希望的替代方案。有了可以估计序列空间适应度景观的计算模型,现在可以选择具有高适应度的候选设计,并采用迭代过程实现生物部件的有效虚拟筛选(图9A)。

图9 A, 生物部件设计的设计与构建-测试-学习(DBTL)流程示意图。生物部件设计将AI引导的虚拟筛选、生物部件合成、生物测量和功能特征学习整合到一个闭环框架中。B, 关于生成模型和预测模型的框架。
从机器学习的角度来看,生物部件的逆向设计问题可以抽象为估计生物部件功能联合分布的数学问题,并从中采样目标生物部件x和目标功能y。在目标功能y的情况下,生物部件设计问题可以以概率形式表述为寻找相互兼容的序列-功能对,以最大化联合概率p(x,y)。使用概率链规则,我们可以得到:p(x,y)=p(y∣x)⋅p(x)其中第一项表示给定序列x的功能y的条件概率,第二项表示受到化学和生物物理属性限制的序列x的生物兼容性。机器学习方法的发展,特别是深度学习,使得适应度环境的估计越来越准确,并显著提高了生成满足生物学约束条件的候选设计的效率。将虚拟筛选和高通量实验筛选结合在一个闭环中,促进了虚拟筛选的迭代优化,并进一步加快了设计进展(图9A)。
10.2.深度学习模型在合成生物学中的应用
随着计算能力的增强和高通量组学数据的增加,深度学习的使用已成为学习复杂模式和隐式或显式高效估计数据分布的有希望的方法。我们简要介绍了在生物组件设计中广泛使用的两类主要深度学习模型:预测模型和生成模型。预测模型:为了估计项p(y/x),构建了预测模型以评估在输入生物部件
来源:药时空 关键词:
合成生物学