嘉峪检测网 2025-04-23 11:32
导读:本文调研了体内基因编辑治疗药品和体外基因编辑细胞药品的研究进展和申报现状,围绕基因编辑酶和sgRNA结构、纯度、体外基因编辑效率、体外编辑特异性等提出基因编辑器药学设计和质量评价的考虑,为该类药品的研发和质量评价提供参考。
[摘要] 近年来基因编辑生物技术药物展现出巨大的临床治疗潜力,随着表观遗传编辑、线粒体基因编辑等先进技术的不断发展,基因编辑器序列和结构也在快速迭代。但该领域现阶段仍面临精准度不足、编辑效率波动大、脱靶效应显著等问题,制约了基因编辑治疗药品临床的广泛应用。基因编辑治疗药品药学研究中,基因编辑器序列设计和结构、纯度等质量属性的差异直接影响该类药品的编辑准确性和编辑精度,进而影响药品的安全性和有效性。基因编辑药品研发和监管领域需要探索合理的多维度质量评估策略,建立科学高效的药品监管工具。本文调研了体内基因编辑治疗药品和体外基因编辑细胞药品的研究进展和申报现状,围绕基因编辑酶和sgRNA结构、纯度、体外基因编辑效率、体外编辑特异性等提出基因编辑器药学设计和质量评价的考虑,为该类药品的研发和质量评价提供参考。
1.前言
基因编辑(genome editing)指在体内或体外采用核酸酶等工具实现序列添加、删除、改变、更换等方式改变人基因组特定位置DNA 序列的过程。近年来,新型基因编辑、碱基编辑、先导编辑、表观基因组编辑和线粒体基因编辑等技术手段的发展日新月异,占据了生物医药产业创新战略高地,创新研发生态日益成熟[1] 。目前全球在研的基因编辑类药品包括体内基因编辑药品(in vivogenome modification)和体外基因修饰细胞(ex vivogenome modification)和体外基因修饰细胞(ex vivogenome modification)两类基因编辑药品。由CRISPR/Vertex 公司联合研发的CRISPR/Cas9 体外基因编辑造血干细胞Casgevy(exagamglogene autotemcel, Exa-cel)已于2023 年11和12 月先后被英国药品和健康产品管理局(MHRA)、美国FDA授予有条件上市许可[2] 。全球首款进入临床试验的基因编辑治疗产品NTLA-2001由Intellia制药公司研发,是现阶段进展最快的体内CRISPR基因编辑疗法。我国基因修饰治疗药品的研发和注册申报数量也在逐年增加,截至2024 年12 月,约有10 余个基因编辑治疗产品获得国家药品监督管理局(NMPA)药品审评中心默示许可进入临床试验(见表1)。
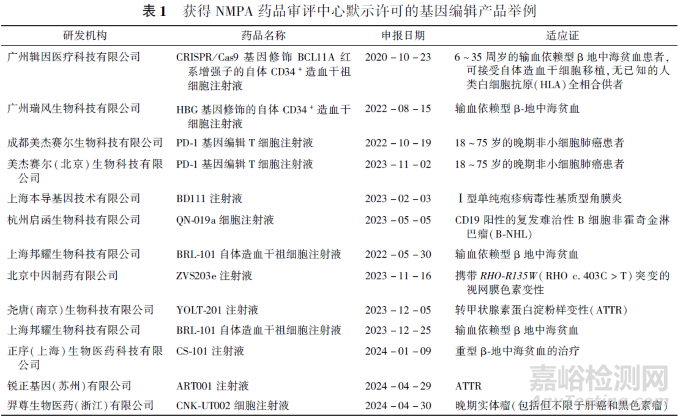
不同基因编辑工具(基因编辑酶、向导RNA等)和其递送系统的组合衍生出多种复杂的基因编辑类药品,其结构设计和质量方面的差异可直接影响到体内基因编辑药品/体外基因修饰细胞的安全性和有效性。现有的基因编辑技术虽已展现出巨大的潜力,但仍面临精准度不足、编辑效率波动大、脱靶效应显著等问题[3] 。产生双链DNA 断裂的基因编辑技术可能产生染色体断裂相关的基因组不稳定的风险,对于儿科患者和育龄患者的体内基因编辑需要充分评估对下一代的影响。不同类型基因编辑工具临床应用过程中,脱靶编辑、非预期在靶编辑和染色体重组等发生的程度也各有差异[4] 。为确保基因编辑的安全、精准和高效,需要研究明确基因编辑药品的生产用原材料、工艺和质量研究的监管考虑,推动质量可控、安全有效的基因编辑药物快速审批上市。本文将介绍该类产品的药学评价(CMC)研究现状和审评考虑,给研发人员提供参考。
2.基因编辑治疗药物CMC研究现状
2.1 基因编辑治疗药品CMC研究挑战
对于使用了基因编辑器的细胞基因治疗产品,现阶段需要采用多种方法对CRISPR-Cas, CBE/ABE碱基编辑酶等编辑系统进行全面的风险分析和评估。例如:编辑系统自身特定的局限性、序列靶向的特异性、脱靶位点的分析、编辑酶的保真性(fidelity)和编辑效率、编辑系统的生产和质量控制、编辑技术对细胞促瘤/成瘤的优势筛选、编辑系统组分的免疫原性、编辑工具相关序列的非特异性插入、多靶点编辑的基因组重排等[5] 。基因编辑工具的编辑准确性(编辑位点的特异性)和编辑精度(即产生确切的所需编辑结果)是目前基因编辑类药品研发和审评领域的关键挑战[6] 。质量研究方面需要结合科学研究进展和方法学的改进,完善基因编辑系统的质控策略。
基因编辑器的序列筛选和结构设计方面,为了降低基因编辑酶发生非预期基因组切割的概率,常见的策略包括通过生物信息学预测模型筛选基因编辑酶序列、结合结构生物学合理优化酶结构以减少非酶依赖的核酸结合、使用新的基因递送方法将基因编辑工具靶向递送到目标位点等[7] 。因此需要结合目标产品的体内外研究数据以及现有生物信息学预测工具,对基因编辑器的准确性、编辑精度进行评价和确认,确认筛选和优化过程的合理性和安全性。
基因编辑药物质量研究方面,考虑到DNA、RNA、蛋白质等不同基因编辑工具的物质基础各不相同,也需要相应对基因编辑工具的结构(宏观/微观)、纯度、生物学活性进行研究,探索各质量属性和临床安全性、有效性的关联,确认关键质量属性的判定依据和标准限度。
然而,基因编辑工具筛选和特异性分析、深度测序等产生大量生物信息学数据,该类数据评价时往往需要借助生物信息学分析工具,并可能需要模拟数据(in silico data)进行方法学验证[8] ;基因编辑工具的质量研究方面,复杂修饰sgRNA的质量分析方法仍在不断探索优化中,序列确认、纯度、序列变异体、二级结构和碱基修饰分析等方法学验证均存在较多难点[9] ;基因编辑细胞终产品的表征中,患者个体基因组和细胞质量等方面的差异可能导致基因编辑效率和编辑精度产生较大差异。综上,基因编辑器序列、结构的多样性和质量变异性需要多维度进行质量评价,为该类药品的科学监管带来了巨大的挑战。
2.2 基因编辑治疗药品CMC技术指南
目前,包括美国FDA、欧洲EMA、日本PMDA 在内的发达国家监管机构均发布了基因编辑药物相关技术指南,提出了基因编辑类产品前沿技术评价考虑[10-11] 。国内外各监管机构发布的指南中含有部分基因修饰系统的药学技术要求,说明需要评估基因编辑产品分子设计、工艺研究、质量研究与质量控制评价方法及标准等[12-13] ,但未针对不同类型基因修饰系统明确技术要求。
研究和评价方面,基因编辑工具筛选相关数据库、预测算法及分析流程、深度测序分析方法及验证等先进工具系统化和标准化的评价方法全球缺乏一定的共识。美国FDA结合自有的基因编辑药物质量和安全性数据库以及产品审评经验积累,对于该类基因编辑器的纯度、编辑精度等部分质量控制项目提出了具体研究方法和标准[14] 。在2022 年12月召开的学术讨论会中,美国FDA提出了需要借助机器学习等先进的工具开展基因编辑产品精准性评价方面的监管研究,并强调建立sgRNA数据库等用于审评标准研究的重要性[15] 。我国也在2022 年发布的《体外基因修饰系统药学研究与评价技术指导原则(试行)》等指南中提出了初步考虑,但尚未针对基因编辑工具和药品发布相关系统性的指南[16] 。在基因编辑酶序列、结构合理设计和关键质量属性评价方面,需要将准确的基因编辑科学知识与药品评价相结合,建立能够准确反映基因编辑工具质量和安全有效性的基因编辑药品评价技术要求。
3. CRISPR-Cas类基因编辑治疗药品质量评价考虑
3.1 序列设计与上游构建
优化基因编辑器的结构是降低脱靶效应的主要途径。目前,有多种优化方法可提高编辑效率,降低脱靶效应和基因组重排,包括sgRNA序列优化、编辑酶结构优化等。近年来,多种人工智能工具逐渐应用于基因编辑器效果预测和优化,通过添加基因编辑领域专业知识和外部工具,进行CRISPR基因编辑、表观遗传编辑、先导编辑和碱基编辑工具的编辑效率和脱靶效果的预测分析[17-19] 。尽管这类人工智能模型具有庞大的数据库,但其缺乏精确的基因编辑药物先验知识和试验细节(如sgRNA纯度、用量等),容易产生不准确的响应。应用生物信息学预测软件筛选的基因编辑器,其药学质量评价也需要结合体内、体外研究确认筛选序列的合理性。
由于序列设计规则和算法不同,不同序列设计软件或平台推荐的sgRNA 等候选靶向结合序列可能会有较大的差异,评价时需要综合对比多个平台的设计和潜在脱靶位点的分析筛选候选序列。sgRNA通常由100 个核糖核苷酸组成,主要包含5'端的spacer序列以及3' 端的骨架序列。建议分析sgRNA序列的loop 结构、高级结构、上下文碱基、碱基修饰等特征,通过计算机分析确认sgRNA结合的DNA 序列除靶基因外,人体其他基因组区域没有同源的DNA 序列,结合体外试验研究脱靶位点,了解脱靶发生的频率和影响。鼓励研究探索序列设计与基因编辑效率、脱靶分析、生物学活性等的相关性,不断优化基因编辑工具分子设计。
3.2 基因编辑工具的质量研究
基因编辑工具相关的质量研究项目包括序列、结构、纯度、体外基因编辑效率、体外基因编辑特异性、生物学活性、一般理化特性和递送系统相关质量研究等。该类产品的质量研究需要选择先进且灵敏度满足分析需求的方法,由于分析方法本身可能存在的局限性,可考虑同时采用原理互补的不同方法进行研究。通过全面的质量研究确认产品的关键质量属性(critical quality attributes, CQA)。体内基因药品生产涉及的基因编辑酶和sgRNA 可能通过脂质纳米粒包裹、腺相关病毒(adeno-associated virus, AAV)载体递送等方式用于人体给药,其质量要求相比于体外基因编辑细胞使用的基因编辑工具更为严格。本文此处围绕体内基因治疗Cas9 蛋白和sgRNA序列的质量研究进行介绍。
3.2.1 Cas9 基因编辑酶质量研究和评价策略
重组表达Cas9蛋白的质量研究可参考重组蛋白类生物制品的药学评价技术指南。对于编码Cas9蛋白的mRNA,药学研究可参考核酸类体内基因治疗产品的要求。考虑到Cas9蛋白序列可能影响基因编辑药品的基因编辑效率和脱靶效率,建议关注mRNA 序列的准确性和完整性。对于生物学活性,由于Cas9基因编辑酶需要结合sgRNA才可定位至靶基因区域发挥基因切割活性,结合产品作用机制,可以和sgRNA一并考察。另外,由于该类产品需要在体内发挥作用,需要关注mRNA 的稳定性是否可以支持体内基因编辑。
3.2.2 sgRNA 质量研究和评价策略
sgRNA的spacer序列由5' 端的20个核苷酸组成,它可通过碱基互补配对的方式识别靶向序列,并将Cas9-sgRNA复合物招募至需要编辑的位点;3' 端骨架序列主要通过参与Cas9蛋白的结合并引起后者的构象改变从而介导其内切酶活性,实现靶位点双链切割。sgRNA结构确证检测项目通常包括分子量检测、序列分析(如二代测序)、碱基修饰鉴定、纯度杂质分析等。
序列分析:sgRNA序列的合成工艺较为复杂,4 种单体碱基的缩合效率差异等原因可导致不同序列的sgRNA合成质量具有差异性。如使用二代测序方法, 考虑到该方法一般通过连接建库构建sgRNA文库,通过二代测序平台检测整个RNA 分子的序列正确性,因此在序列正确性分析方法学研究过程中,需要关注样品前处理操作的合理性、序列扩增均匀性、测序文库的质量、测序的准确性和重复性,相应建立二代测序方法和结果评价策略。
纯度和杂质:sgRNA的纯度对该类产品基因编辑的精度和准确度具有较大影响[20] ,进而影响该类产品的有效性和安全性。sgRNA相关杂质包括不同长度(超长序列、截短序列)、不同修饰类型(硫代磷酸立体化学异构体、脱硫序列、脱碱基序列)、支链杂质等,这些杂质可能增加产品的脱靶风险,可考虑采用LC⁃MS、毛细管电泳、离子层析或反向色谱等不同原理的方法开展产品相关杂质的研究[21] 。
为了提高sgRNA在体内的稳定性以及介导的基因编辑的效率并同时降低脱靶的频率,现阶段常见sgRNA序列中引入了不同的修饰位点,主要包括核苷酸的2'⁃O⁃甲基修饰以及核苷酸硫代修饰等。sgRNA的碱基修饰显著影响CRISPR基因编辑系统的准确性和特异性。据报道,采用N6-methyladenosine, 5-methylcytidine和pseudouridine修饰可显著提升sgRNA 与靶序列的配对紧密性,提升Cas9酶对目标DNA 效率的切割效率和基因编辑效率,同时降低sgRNA的免疫刺激和细胞毒性,建议研究时进一步设计相应的实验验证[20] 。
3.3 基因编辑细胞的质量研究
在常见细胞治疗药品的质量研究内容基础上,基因编辑细胞特有的质量研究项目包括体外基因编辑效率、基因修饰细胞数量、体外基因编辑特异性和染色体重排分析等。当使用病毒载体、脂质纳米粒等工具将基因组编辑工具引入细胞时,需要从基因转导工具的感染性和细胞趋向性的角度分析目的细胞和非目的细胞基因修饰的程度和频率,还应考虑到基因组编辑酶的持续表达增加脱靶效应的可能性。由于病毒载体介导的基因组编辑会导致基因组编辑酶的长期持续表达,建议评估基因组编辑酶的持续表达可能产生的后果。根据现阶段基因编辑细胞药物的研究现状,基因编辑细胞的研究通常包括体外基因编辑效率、体外基因编辑特异性、体外成瘤性、生物学活性等药学专业相关内容,也包括脱靶分析、体内药效研究等非临床研究关注的内容。基于基因编辑细胞药物的药学审评经验和相关数据,本文重点介绍体外基因编辑效率、体外编辑特异性的药学审评考虑和多靶点基因编辑质量评价的特殊考虑。
3.3.1 体外基因编辑效率研究
基因编辑效率检测包括采用编辑后细胞样品进行测序(一代测序或二代测序),或基于PCR扩增和测序的方法进行基因编辑效率评价。该方法涉及靶向基因组序列的PCR扩增、一代测序分析、二代测序及生物信息学分析、基因编辑效率计算方式的评价。考虑到该分析方法使用生物信息学方法,需要关注基因编辑效率检测方法的基线噪声、测序深度、测序深度变异系数、检出Indel类型数、生物信息学软件参数的合理性。该方法评价难点在于,CRISPR-Cas基因编辑细胞中基因编辑结果呈现出序列的多样性,需要关注检测结果的准确性和基因编辑效率计算方法的合理性。例如:考虑到细胞基因组序列的多样性,需开展基因编辑效率检测方法在多种序列混合样品中检测结果的准确性验证研究,合理拟定质量标准限度。
3.3.2 体外基因编辑位点特异性研究
当前较多产品采用GUIDE-SEQ,CIRCLE-SEQ和WGS进行编辑特异性预测(分析人基因组中与靶向位点序列高度相似的序列)和特定位点扩增测序检测,评估基因编辑工具的编辑特异性。在编辑位点特异性评价方面需要筛选出潜在脱靶突变位点上下游20 bp内带有5'-NGG-3' PAM的序列,考虑基因非特异编辑主要由于sgRNA与dsDNA靶点序列之间存在数目不定的错配,来假设sgRNA-dsDNA之间同时存在的碱基错配个数(mismatch),进而过滤序列。在方法的评价过程中需要关注碱基错配个数设置的合理性、建库流程、扩增子测序深度和本底变异水平、假阳性脱靶位点、生物信息方法学等方面的研究和验证。
生物信息学预测的方法通过在人类全基因组数据范围内比对和鉴定与sgRNA靶位点相似的DNA序列,进而预测潜在的错配位点,然后再通过扩增子深度测序确认预测的脱靶位点是否真实存在。需要关注检出的错配是样品本身遗传背景导致,还是基因编辑工具特异性不足导致[22] 。目前公开的基因组数据库中,亚洲人群的基因组数据相对较少。基于白人的数据库检测出的错配可能与我国患者的实际情况存在差异,需要探索分析不同人种和不同来源的比对基因组对基因编辑位点特异性评价的影响,确定适用于中国人群的比对基因组的来源。对于遗传多样性高的人群,如具有非洲血统的人群中拷贝数变异和重复区域更难评估,需要获得更多基因组序列信息进行更充分的遗传多样性分析[23-24] 。
3.3.3 多靶点基因编辑质量评价的特殊考虑
在多靶点基因编辑过程中,考虑到基因组中2 个及以上“双链DNA 断裂”的形成可能产生染色体易位风险。审评建议针对性开展分析研究,例如:易位风险一般可采用引物延伸介导测序(Primer extension mediated sequencing, PEM-seq),长距离PCR(long range PCR)等方法分析评估,对于染色体畸变可考虑采用G 带分析、多色荧光原位杂交(mFISH)、比较基因组杂交(CGH)等进行分析。但是这些分析方法也存在较多限制,例如:G 带分析很难同时处理多个细胞并检测到极少数具有染色体畸变的细胞。在发生“同源重组修复”(HDR)的基因编辑细胞中也报道了p53基因发生突变的情况,并且在敲除p53基因的细胞中HDR 效率增加,对于该类情况质量研究中审评也建议考虑关注p53基因突变细胞等相关特征的检测[25]。
4.结语
近年来基因编辑技术发展迅速,药物开发领域的应用也逐渐增多,多个临床试验药物在肿瘤、血液病、神经肌肉等多种适应证中获得了临床数据证据。随着技术的进步,研发者开始探索研发如多靶点基因编辑、不同基因编辑工具的组合使用等更为复杂的基因编辑药品。该类产品的作用原理复杂、原材料种类多样、生产工艺和质量研究的药学评价存在较大难度。质量评价过程中也需要结合临床适应证和产品特点进行风险评估,并需要积累生物信息学等前沿技术领域的科学认知,结合药理毒理和临床多专业考虑设定合理的评价策略。监管科学研究也可尝试通过使用人工智能工具,结合药品审评数据积累,形成基因编辑产品质量评价标准,通过发布相关标准或技术共识文件,高效协同提高相关产业创新发展能力和竞争力。期望通过监管机构、学术界和工业界的合作,加强与国际同行的沟通交流,促进我国基因编辑行业健康有序发展,同步提升我国先进治疗药品的监管水平。
参考文献
[1] DOUDNA JA. The promise and challenge of therapeutic genomeediting[J]. Nature, 2020, 578(7794): 229 -236.
[2] PARUMS DV. Editorial: first regulatory approvals for CRISPR⁃Cas9 therapeutic gene editing for sickle cell disease and transfu⁃sion⁃dependent
[3] KIM D, LUK K, WOLFE SA, et al. Evaluating and enhancingtarget specificity of gene⁃editing nucleases and deaminases[J]. Annu Rev Biochem, 2019, 88: 191 -220.
[4] LI HY, YANG Y, HONG WQ, et al. Applications of genomeediting technology in the targeted therapy of human diseases:mechanisms, advances and prospects[J]. Signal Transduct Tar⁃get Ther, 2020, 5(1): 1.
[5] MADDALO D, MANCHADO E, CONCEPCION CP, et al. Invivo engineering of oncogenic chromosomal rearrangements with theCRISPR/ Cas9 system[J]. Nature, 2014, 516 (7531): 423 -427.
[6] EISENMAN D, SWINDLE S. Food and drug administrationguidance on design of clinical trials for gene therapy products withpotential for genome integration or genome editing and associatedlong⁃term follow⁃up of research subjects[J]. Appl Biosaf, 2022,27(4): 201 -209.
[7] TAN JJ, ZHANG F, KARCHER D, et al. Engineering of high⁃precision base editors for site⁃specific single nucleotide replace⁃ment[J]. Nat Commun, 2019, 10(1): 439.
[8] CHEN L, LIU GQ, ZHANG T. Integrating machine learning andgenome editing for crop improvement[J]. aBIOTECH, 2024, 5(2): 262 -277.
[9] WEI BC, WANG J, DAI LL, et al. Characterization of syntheticguide ribonucleic acids through hydrophilic interaction chromatog⁃raphy coupled with mass spectrometry [ J]. J Chromatogr A,2023, 1710: 464414.
[10] FDA. 2020, Chemistry, Manufacturing, and Control (CMC) In⁃formation for Human Gene Therapy Investigational New Drug Ap⁃plications (INDs) [EB/ OL]. (2023). https:/ / www. fda. gov/regulatory⁃information/ search⁃fda⁃guidance⁃documents/ chemistry⁃manufacturing⁃and⁃control⁃cmc⁃information⁃human⁃gene⁃therapy⁃investigational⁃new⁃drug.
[11] European Medicines Agency. Guideline on quality, non⁃clinicaland clinical aspects of medicinal products containing geneticallymodified cells[EB/ OL]. (2020). https:/ / www. ema. europa.eu/ en/ quality⁃non⁃clinical⁃clinical⁃aspects⁃medicinal⁃products⁃containing⁃genetically⁃modified⁃cells.
[12] FDA. Human Gene Therapy Products Incorporating Human Ge⁃nome Editing, Guidance for Industry [ EB/ OL]. (2024). ht⁃tps:/ / www. fda. gov/ regulatory⁃information/ search⁃fda⁃guidance⁃documents/ human⁃gene⁃therapy⁃products⁃incorporating⁃human⁃ge⁃nome⁃editing.
[13] TAVRIDOU A, ROGERS D, FARINELLI G, et al. Genome⁃ed⁃iting medicinal products: the EMA perspective [ J]. Nat RevDrug Discov, 2024, 23(4): 242 -243.
[14] FDA CBER. Webinar: Human Gene Therapy Products Incorpora⁃ting Human Genome Editing[EB/ OL]. (2024 - 02 - 29). ht⁃tps:/ / www. fda. gov/ regulatory⁃information/ search⁃fda⁃guidance⁃documents/ human⁃gene⁃therapy⁃products⁃incorporating⁃human⁃ge⁃nome⁃editing.
[15] FDA. CBER Webinar: workshop: Assessing Genetic Heterogene⁃ity in the Context of Genome Editing Off⁃Targets in Gene TherapyProducts[ EB/ OL]. (2022). https:/ / www. fda. gov/ vaccines⁃blood⁃biologics/ workshops⁃meetings⁃conferences⁃biologics/ asses⁃sing⁃genetic⁃heterogeneity⁃context⁃genome⁃editing⁃targets⁃gene⁃therapy⁃products⁃12162022.
[16] 国家药品监督管理局药品审评中心. 国家药监局药审中心关于发布《体外基因修饰系统药学研究与评价技术指导原则(试行)》的通告(2022 年第29 号) [EB/ OL]. (2022). ht⁃tp:/ / assist1. cdeapp. org. cn/ office/ officeWorkNews/ show? code⁃Wid =7d747bcaa596432ba7952e07d147e1fb&wtype =2.
[17] DIXIT S, KUMAR A, SRINIVASAN K, et al. Advancing ge⁃nome editing with artificial intelligence: opportunities, challen⁃ges, and future directions[J]. Front Bioeng Biotechnol, 2024,11: 1335901.
[18] ARBAB M, SHEN MW, MOK B, et al. Determinants of baseediting outcomes from target library analysis and machine learning[J]. Cell, 2020, 182(2): 463 -480, e30.
[19] MATHIS N, ALLAM A, KISSLING L, et al. Predicting primeediting efficiency and product purity by deep learning[J]. NatBiotechnol, 2023, 41(8): 1151 -1159.
[20] PROKHOROVA DV, VOKHTANTSEV IP, TOLSTOVA PO, et al.Natural nucleoside modifications in guide RNAs can modulate theactivity of the CRISPR⁃Cas9 system in vitro [ J]. CRISPR J,2022, 5(6): 799 -812.
[21] BECETTE OB, TRAN A, MARINO JP, et al. Rapid identifica⁃tion of short oligonucleotide impurities using lithium adduct con⁃solidated MALDI⁃TOF mass spectrometry[J]. Int J Mass Spec⁃trom, 2022, 481: 116913.
[22] SCOTT DA, ZHANG F. Implications of human genetic variationin CRISPR⁃based therapeutic genome editing [ J]. Nat Med,2017, 23(9): 1095 -1101.
[23] CANCELLIERI S, ZENG J, LIN LY, et al. Human genetic di⁃versity alters off⁃target outcomes of therapeutic gene editing[J]. Nat Genet, 2023, 55(1): 34 -43.
[24] CANVER MC, KEITH JOUNG J, PINELLO L. Impact of geneticvariation on CRISPR⁃cas targeting[J]. CRISPR J, 2018, 1(2):159 -170.
[25] HAAPANIEMI E, BOTLA S, PERSSON J, et al. CRISPR⁃Cas9genome editing induces a p53⁃mediated DNA damage response[J]. Nat Med, 2018, 24(7): 927 -930.
内容来源:中国新药杂志2025 年第34 卷

来源:Internet
关键词: 基因编辑治疗药品